蚕豆遗传图谱与QTL定位研究进展
周仙莉 滕长才 张红岩 林夕 刘玉玲 吴小燕 侯万伟 刘玉皎
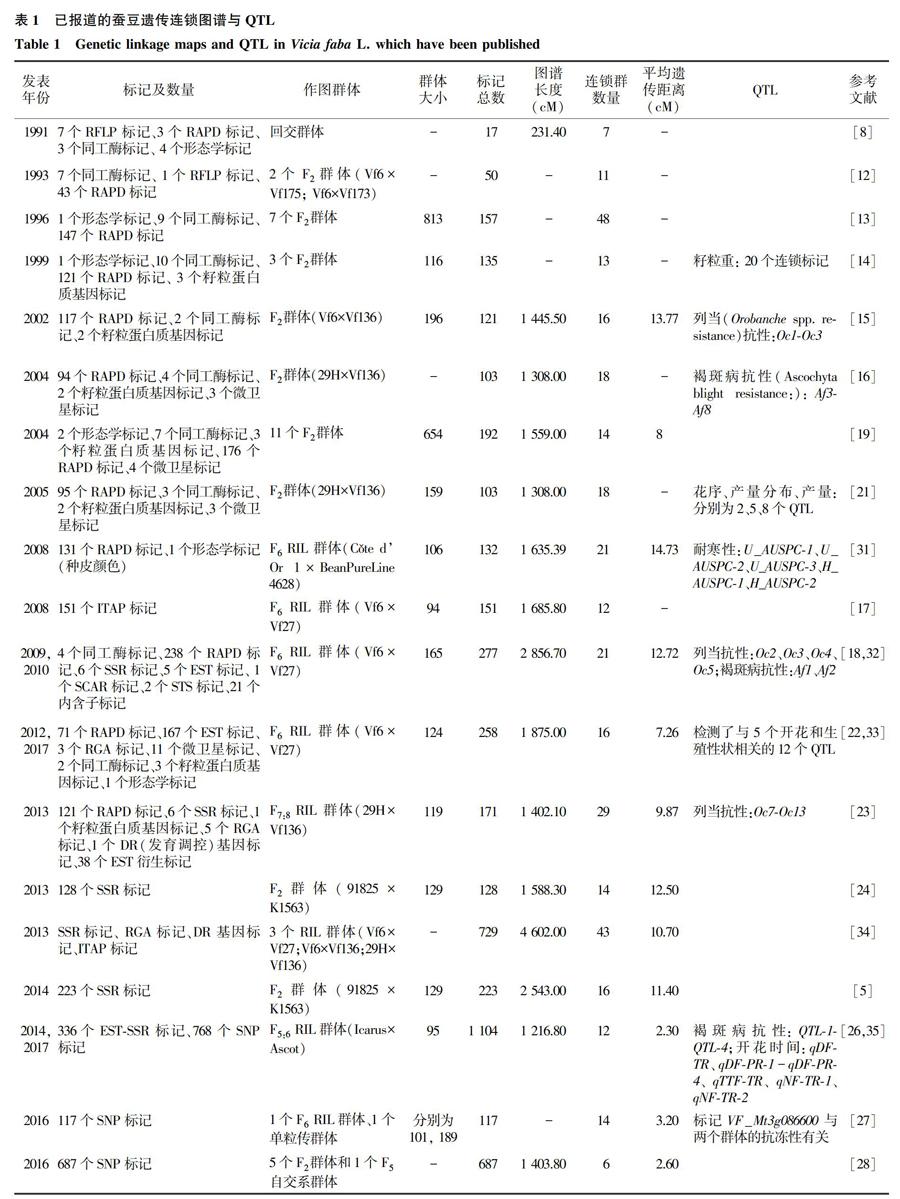
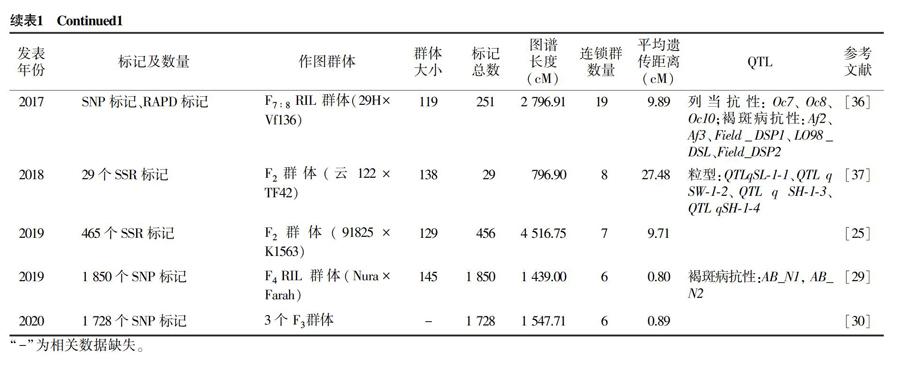
摘要: 作为一种粮食、蔬菜、饲料、绿肥兼用的食用豆类,蚕豆在人类健康、土壤改良等方面发挥了重要作用。高密度遗传图谱的构建是蚕豆基因组研究的基础和主要手段,至今共构建了24张蚕豆遗传图谱,最为饱和的图谱覆盖长度为1 439 cM,平均遗传距离为0.8 cM。同时基于图谱开展了产量相关性状、品质相关性状以及抗性相关性状的QTL定位,有效地促进了蚕豆的遗传与基因研究。
关键词: 蚕豆;遗传连锁图谱;QTL定位
中图分类号: S529 文献标识码: A 文章编号: 1000-4440(2021)01-0237-09
Research advance of genetic linkage map and QTL location in Vicia faba L.
ZHOU Xian-li1,2, TENG Chang-cai2, ZHANG Hong-yan1,2, LIN Xi1,2, LIU Yu-ling1,2, WU Xiao-yan1,2,HOU Wan-wei2, LIU Yu-jiao1,2,3
(1.Qinghai University, Xining 810016, China;2.Qinghai Academy of Agricultural and Forestry Sciences, Xining 810016, China;3.The Co-constructing State Key Laboratory of Three Rivers Sources Ecology and Plateau Agriculture and Animal Husbandry, Qinghai University, Xining 810016, China)
Abstract: As a kind of edible legumes which can be used as food, vegetables feed and green manure, Vicia faba L. plays a crucial role in human health and soil improvement. The construction of high-density genetic linkage map is the basis and main method for genome research in V. faba L.. 24 genetic maps have been constructed so far, in which the most saturated map spanned 1 439 cM with an average genetic distance of 0.8 cM. Quantitative trait locus (QTL) mapping on yield-related traits, quality-related traits, and resistance-related traits had been carried out, which effectively promoted inheritance and gene research in V. faba L..
Key words: Vicia faba L.;genetic linkage maps;quantitative trait locus (QTL) location
蠶豆(Vicia faba L.)具有“养人、养畜、养地”的作用,是中国传统的出口农产品,也是青海省种植业结构调整和农民增收的优势作物之一[1-3]。随着人们生活水平的提高和健康饮食需求的增加,菜用蚕豆的市场需求量越来越大。蚕豆作为一种轮作豆类,在土壤改良方面也发挥着重要作用。
遗传连锁图谱是指已知基因或分子标记在连锁群上的线性排列,标记间的相对遗传距离通过重组率计算[4-5]。蚕豆基因组庞大(约13 Gb),缺乏可用的基因组序列数据,导致其基因组研究进展缓慢[6]。构建高密度遗传图谱,进行QTL定位以及开发连锁标记是蚕豆基因组研究的主要手段,对控制重要性状的关键基因定位与克隆、关联与连锁分析以及分子标记辅助选择优良品种具有重要作用。
基于蚕豆遗传图谱,对产量、品质、抗性等重要性状进行基因与QTL定位,并针对部分性状开发连锁标记,有利于替代繁琐且耗时的表型评估,促进分子标记辅助选择(MAS)育种的发展,进而缩短育种进程[7]。本文对蚕豆遗传图谱的构建与重要农艺性状、品质性状以及抗性的QTL定位的研究进展进行综述,旨在为蚕豆遗传连锁图谱的加密、重要性状的基因精细定位和MAS育种等提供参考。
1 蚕豆遗传连锁图谱的构建
蚕豆遗传连锁图谱的构建起源于1991年,Van de Ven等[8]报道了蚕豆第1张遗传图谱。在随后的近30年里,基于12种遗传标记共构建了24张遗传图谱(表1)。蚕豆遗传图谱的研究进程可大致分为以下2个阶段:第1阶段是利用形态学(Morphological markers)、同工酶(Isozymes)等遗传标记与随机扩增多态性(Random amplified polymorphic DNA,RAPD)标记、微卫星(Microsatellite)等传统标记构建蚕豆遗传图谱;第2阶段是利用高通量测序技术开发的表达序列标签(Expressed sequence tag,EST)、单核苷酸多态性(Single nucleotide polymorphisms,SNP)等标记构建蚕豆遗传图谱。
1.1 基于形态学、RAPD等标记的蚕豆遗传图谱
早期,国外学者对种皮颜色等形态学性状与一些同工酶系统进行了报道,并研究了其基因在染色体上的位置[9-11]。这些形态学性状和同工酶系统被作为遗传标记广泛应用于蚕豆遗传图谱的构建[8, 12-14]。基于同工酶、限制性片段长度多态性(Restriction fragment length polymorphism,RFLP)等传统标记构建的遗传图谱的标记数量较少,密度较低。Van de Ven等[8]构建的第1张图谱仅包含7个标记;Romn等[15]构建的遗传图谱包含121个标记,平均遗传距离为13.77 cM。构建的蚕豆遗传图谱包含的连锁群数量多于蚕豆染色体数(2n=12)。Satovic等[13]构建的图谱包含了48个连锁群,而仅有6个连锁群被锚定到了染色体上。
在作图群体构建上,常以Vf6[12-15]、Vf136[13,15-16]、Vf27[17-18]等品系作为亲本,构建回交群体(Backcross population,BC population)[8]、F2群体以及重组自交系(Recombinant inbred lines,RIL)等群体作为作图群体以构建图谱。以蚕豆三体材料为亲本构建作图群体,可以将连锁群与染色体相关联。蚕豆初级三体是以Vf6为母本与其他材料雜交所形成的,用于检测建立遗传图谱中连锁群与染色体之间的对应关系[13-14]。Satovic等[13]将Vf6分别与不同蚕豆染色体三体材料杂交,形成了具有3、4、5、6号染色体三体的7个F2作图群体,所构建的图谱包含1个形态学标记、9个同工酶标记与147个RAPD标记。由于单一群体间多态性有限,所以基于多个作图群体构建了复合图谱,锚定了更多的遗传标记,增加了图谱密度[13-14,19]。
2002年,Poarkova等[20]从蚕豆特异性DNA文库中开发的位于蚕豆1号染色体上的微卫星标记也被广泛应用于蚕豆遗传图谱的构建[16,19,21]。Ellwood等[17]则利用RIL群体构建了包含151个内含子靶向扩增多态性(Intron-targeted amplified polymorphic,ITAP)标记、12个连锁群、覆盖1 685.8 cM的遗传图谱,每个连锁群上的标记数量由3个到30个不等,长度从23.6 cM到324.8 cM不等,与蒺藜苜蓿染色体组之间的同源分析结果表明两种作物之间具有高度的共线性。
1.2 基于高通量测序技术的蚕豆遗传图谱
随着分子标记和高通量测序技术的不断发展,EST衍生标记[22-23]、SNP标记开始被应用于蚕豆遗传图谱的构建。为了对蚕豆开花与产量性状进行QTL定位,Cruz-Izquierdo等[22]基于Vf6×Vf27衍生的RIL群体构建了具有16个连锁群、258个标记的遗传图谱,其中EST标记167个,其他标记则为同工酶、RAPD等标记,并将构建的遗传图谱与蒺藜苜蓿和兵豆的染色体进行了共线性分析,为建立蚕豆基因组与其他豆科作物基因组之间的同源性提供了锚定位点。自2013年起,国内开始采用EST(表达序列标签)序列设计和已发表的简单重复序列(Simple sequence repeats,SSR)标记构建蚕豆遗传连锁图谱,并对图谱进行加密[5,24-25]。在Ma等[24]构建的SSR遗传图谱基础上,Yang等[25]继续以91825×K1563构建的F2群体为作图群体进行加密,最终构建了包含465个SSR标记、7个连锁群、覆盖长度为4 516.75 cM、平均遗传距离为9.71 cM的遗传图谱,连锁群上的SSR标记数量从12到136不等,连锁群的长度范围为129.35 cM至1 180.21 cM,图谱覆盖度极大提升,但平均遗传距离仍较大。
2014年起,SNP标记开始被应用于蚕豆遗传图谱构建[26-28]。至今最饱和的蚕豆遗传图谱是由Sudheesh等[29]构建的整合图谱,Sudheesh等基于SNP标记将已发表的遗传图谱与其研究中新构建的遗传图谱相整合,构建了一张包含6个连锁群、1 850个标记的整合图谱,其覆盖总长度为1 439 cM,平均遗传距离为0.8 cM。最近,Carrillo-Perdomo等[30]利用在3个重组品系中表现多态性的1 819个SNP标记构建了一张包含1 728个标记、图谱长度为1 547.71 cM、平均遗传距离为0.89 cM的高密度蚕豆遗传图谱。
分子标记与遗传图谱的构建是蚕豆基因组研究中的有效手段。目前,RAPD、SSR、SNP等多种不同类型的遗传标记被应用于图谱构建,所构建的蚕豆遗传图谱覆盖长度增加,标记数量增多,并可对多个图谱进行有效整合。饱和遗传连锁图谱的构建和与目标性状紧密连锁的分子标记的开发,对MAS育种中育种进程的缩短具有重要意义。在构建的遗传图谱中,13张遗传图谱被应用于蚕豆产量与籽粒相关性状、开花特性、抗性等重要性状的QTL定位。
1.3 与其他豆科作物的同源比对
将蚕豆遗传图谱与其他豆科作物进行同源比对,以确定蚕豆与其他作物在系统发育中的亲缘关系,是对控制重要性状的关键基因进行同源克隆与候选基因筛选的主要手段之一。将应用来源于其他豆科作物的通用性引物构建的蚕豆遗传图谱与小扁豆、蒺藜苜蓿的基因组进行同源比对,结果表明3种作物间均存在高度的共线性,蚕豆与小扁豆之间的同源水平更高;与蒺藜苜蓿的染色体比较,发现蚕豆和小扁豆的染色体均存在中等程度的重排,这种重排可以解释不同作物间染色体数目的差异[17, 22]。将基于测序技术构建的蚕豆遗传图谱与以上2种作物进行同源比对,发现蚕豆和蒺藜苜蓿基因组间存在大量的高度共线性区域,并在映射区间内通过序列比对验证了1个控制单宁的候选基因[28]。因此,同源比对有助于在具有高度共线性的映射区间内预测基因含量与距离,进行基因注释。
不同作物间的共线性程度在一定程度上反映了作物在系统发育中亲缘关系的远近。蚕豆与蒺藜苜蓿、鹰嘴豆、豌豆等亲缘关系较近,因此在同源比对时常将蚕豆与这些豆科作物进行比较。Kaur 等[26]与Carrillo-Perdomo等[30]均将其所构建的图谱与多种豆科作物的基因组进行了比较。Carrillo-Perdomo等[30]发现蚕豆遗传图谱中的连锁群与豌豆、鹰嘴豆、蒺藜苜蓿等近缘作物染色体之间的共线性区域几乎覆盖了整个染色体组,而蚕豆与菜豆、短豇豆、百脉根之间的亲缘关系较远,故其染色体间存在的共线性区域稍少。
2 蚕豆重要性状相关QTL研究进展
2.1 重要农艺性状与产量相关QTL
产量是蚕豆育种中关注的主要问题,单株荚数、单荚粒数以及百粒质量共同构成了蚕豆单株产量[31-38]。产量形成过程受到多个性状的影响,如开花时间、生长习性等。开花时间主要影响蚕豆品种的成熟特性和环境适应性。Cruz-Izquierdo等[22]鉴定并验证了控制开花时间、花期长度、荚长、单荚粒数、单荚胚珠数等5个性状的QTL,在2、5、6号染色体上连续2年鉴定出12个稳定的QTL,比较分析结果表明控制开花时间的基因组区域在其他豆科作物中具有保守性。Avila等[33]对花序特征、产量相关的8个性状进行了QTL定位,检测到大量的QTL位点,在开花节位、结荚高度、有效分枝数方面各检测到4个QTL,在单花序花蕾数、百粒质量方面各检测到5个QTL,在主茎开花层数方面检测到6个具有加性效应的QTL,但QTL的准确性和稳定性需进一步验证。
百粒质量和粒型性状是重要的商品经济特征。1999年,与百粒质量相关的QTL被首次报道[14],QTL主要聚集在6号染色体上,与20个标记位点显著相关,主效QTL位点(与OPM1811725标记连锁)可解释将近30%的表型变异率。田莹莹等[37]以粒型差异较大的云122与TF42为亲本配置杂交组合,构建F2群体对粒型性状进行了QTL定位,最终检测到4个控制籽粒长、宽、质量的QTL(qSL-1-1、 qSW-1-2 、qSH-1-3、qSH-1-4)。
目前,产量方面的QTL研究较少,定位到的QTL区间较大,其稳定性与准确性需要进一步验证,难以开发可直接应用于MAS育种的分子标记。对于质量性状,如子叶颜色和生长习性,连锁标记的开发难度较低。沙伟超[39]采用分组混合分析(BSA)法筛选到9个与子叶颜色相连锁的SSR标记,并初步将控制子叶颜色的基因定位到LG05,但未对连锁标记进行验证。Avila等[40-41]开发了用于有限生长习性选择的酶切扩增多态性序列(Cleaved amplified polymorphic sequences,CAPS)标记Vf_TFL1,在MAS育种中具有实用性。
2.2 品质性状相关标记
蚕豆营养价值高, 富含蛋白质、淀粉和微量元素[42]。蚕豆蛋白质是一种优质的谷物蛋白质,蛋白质组分含量不同阶段处于不断变化中[43]。Macas等[44-45]报道了5个控制蚕豆籽粒蛋白質的基因,并公布了相关引物序列。蚕豆籽粒中抗营养因子的存在降低了蛋白质的生物学价值,在育种进程中选育抗营养因子含量较低的品种为品质育种提供了新的研究方向[46]。Gutierrez等[47-49]鉴定了与单宁缺失基因(zt-1、zt-2)紧密连锁的2个SCAR标记,报道了与蚕豆嘧啶葡糖苷和伴蚕豆嘧啶核苷含量相关基因紧密连锁的1个CAPs标记。Hou等[50]利用596个SSR标记和100个ISSR标记筛选到1个与zt-1紧密连锁的SSR标记(SSR84),可以准确地预测zt-1基因型。
2.3 抗性相关QTL和基因定位
病害是蚕豆产量的主要限制因素之一,全世界至2018年为止报道了锈病、褐斑病、霜霉病等10种蚕豆常见真菌病害[51]。抗性的复杂性导致难以仅仅依据表型观测进行抗性育种,因此有必要对抗性的遗传机制进行研究。目前,针对锈病、褐斑病、耐寒性、抗旱性的相关QTL进行了研究,赤斑病、霜霉病等病害的遗传研究基础较薄弱,仅见病原鉴定、防治措施及遗传机制等方面的研究报道[52-54]。
2.3.1 锈病与褐斑病抗性 蚕豆锈病和褐斑病是真菌病害,对中国秋播型蚕豆危害严重[55]。典型褐斑病发生时,会造成35%~40%减产[56]。抗病育种过程较为复杂,已育成的抗病品种由于病菌生理小种的变化也易丧失抗性,目前难以获得完全抗性。Sillero等[57]在648份蚕豆种质中筛选出6份(V-300、V-1271、V-1273、V-313、V-1272、V-1335)具有锈病抗性的资源。基于抗锈病品系 2N52和敏感品系VF-176杂交产生的F2群体, Avila等[58]采用分组混合分析法检测到了3个与抗锈病基因(Uvf-1)紧密连锁的RAPD标记,在标记OPI20900和Uvf-1间未检测到重组,采用该标记对抗锈病基因型的选择效率较高。
蚕豆褐斑病抗性的等位基因多来源于Vf6、29H、Ascot等品系,在蚕豆抗褐斑病相关QTL定位中较多地使用了这些品系。初期,基于Vf6×Vf136和29H×Vf136衍生的群体,Román等[56]检测到的控制蚕豆褐斑病抗性的QTL (Af1)与Avila等[16]检测到的Af3都位于3号染色体,表明二者可能位于同一基因组区域。基于Icarus× Ascot衍生的RIL群体,Kaur等[26]在2年的评估中检测到4个相关QTL,分别位于Chr-I.A、Chr-II与Chr-VI,其中,3号位点可能在先前的研究中被报道过,而其他3个则是新检测到的位点。最近,Sudheesh等[29]构建了一张高密度的整合图谱用于蚕豆褐斑病抗性相关QTL定位,作图群体为Nura×Farah衍生的RIL,Nura是从 Icarus和Ascot杂交产生的系谱中选择出来的,其抗性主要来源于Ascot,最终确定了2个基因组区域(AB_N1、AB_N2),可解释多达49%的表型变异,2个QTL与Kaur等[26]检测到的1号位点和4号位点为相同的位点。
2.3.2 列当抗性 在地中海沿岸,列当对于蚕豆的寄生极其严重,难以有效控制[46]。Díaz-Ruiz等[59-60]基于Vf6×Vf136的RIL群体检测到了4个相关QTL区域(Oc2、Oc3、Oc4、Oc5),其中Oc4、Oc5被定位在1号染色体。Díaz-Ruiz等[32]在不同的世代和环境中对列当抗性相关QTL区域进行了验证,最终在不同环境中的3个RIL群体中鉴定出4个控制列当抗性的QTL,而先前报道的在F2群体中检测到的Oc1在高代群体中则不显著。Ocaa-Moral等[36]基于SNP标记对Gutiérrez等[23]检测到的QTL进行了验证,发现在6号染色体上检测到的Oc7在不同年份中均解释了较大的表型变异,并在QTL区间内增添了2个SNP标记,减小了标记间隙,而Oc8位点仅在1年中被检测到。
2.3.3 非生物抗性 MAS育种比常规育种更快速、更有效,尤其是对于性状表达受环境影响较大的非生物胁迫而言[61]。蚕豆的耐寒性是保证秋播型蚕豆安全越冬的前提。基因型和环境的相互作用降低了耐寒性选择的有效性,传统育种程序对于秋播型种质的遗传改良进展缓慢[62]。Arbaoui等和Sallam等基于SNP构建的遗传图谱检测到与耐寒性和耐寒性生理相关的多个重要的推定QTL[31,63]。然而,推定的QTL无法直接应用于分子标记辅助选择育种。随后,Sallam等[64]使用101个RIL和189个不同基因型的单粒传后代作为遗传背景,采用QTL定位和全基因组关联分析,检测到5个与耐寒性和脂肪酸组分相关的SNP位点,经验证在2个群体中有1个SNP标记(VF_Mt3g086600)与抗冻性相关基因紧密连锁,这个标记在先前研究中被证明与产量性状相关。
蚕豆在其生长过程对于干旱反应相对敏感,开花期和结荚期的干旱胁迫会导致蚕豆的严重减产[65]。Khazaei等[66]以211个RIL为作图群体,应用来自蒺藜苜蓿的SNP检测到15个与气孔特性相关的推定QTL,并利用蚕豆与蒺藜苜蓿之间的共线性关系鉴定了位于蚕豆2号染色体上的8个QTL(qSD-2013-1、qSD-2014、qSL-2013、qSL-2014、qGS-2013、qCT-2012、qCT-2013、qCT-2014),这些QTL可能是与抗旱性相关的候选基因。
3 问题与展望
3.1 蚕豆遗传连锁图谱的构建
高质量遗传连锁图谱的构建和与目标性状紧密连锁的分子标记的开发,对MAS育种中育种效率的提高具有重要意义。随着分子标记技术的发展,大量可转移的EST、SNP等标记被用于构建整合图谱,在原有图谱的基础上增添新的标记,极大地增加了图谱的分辨率。高质量的蚕豆遗传图谱也被用于与其他近缘豆科植物模型进行同源比对[26,30]。然而。与小麦、大豆等主要农作物相比[67-68],蚕豆遗传连锁图谱仍未达到饱和。
在蚕豆遗传图谱构建方面主要存在以下问题:①应用于图谱构建的作图群体较小,单一群体内单株数量均在200株以下,影响了遗传图谱的精度。②已报道的蚕豆遗传图谱大多基于传统标记,基于SNP标记构建的遗传图谱仅有6張,开发的易于转移的标记数量较少;③已构建的高密度遗传连锁图谱的总长度多在1 500 cM左右,难以覆盖蚕豆庞大的基因组。部分基于SSR及其他标记的遗传图谱虽然总长度较大,但分辨率不足。
因此,应通过扩大作图群体,开发易转移的EST、SNP等标记以及对已发表的图谱进行整合等方式,进一步促进蚕豆遗传图谱的饱和。将所构建的图谱与已发表的图谱进行整合,是增加图谱饱和度的有效手段之一。同时由于整合图谱包含了来自其他图谱的标记,也有利于对不同研究中检测到的QTL进行比较鉴定,以筛选在多个环境中稳定表达的QTL;其次,蚕豆饱和遗传图谱的构建需要更多的基因组信息,如EST测序信息、细菌人工染色体文库等,以进一步开发更多易于转移的标记,增加图谱饱和程度,提高与重要性状相关的QTL区域的分辨率。
3.2 蚕豆重要性状相关QTL检测
基于遗传图谱,大量的QTL被鉴定,涉及开花特性、产量、粒型、子叶颜色、籽粒蛋白质含量、锈病抗性、褐斑病抗性、列当抗性、耐寒性、抗旱性等多个重要性状,开发了与控制有限生长习性、单宁缺失、锈病抗性、耐寒性等基因紧密连锁的分子标记。
在蚕豆重要性状相关QTL定位方面,存在以下问题:①QTL定位研究多集中于抗性领域,产量方面的QTL报道较少,其稳定性和准确性需进一步验证;②蚕豆遗传图谱的饱和度极大地限制了重要性状的基因或QTL的精细定位和克隆[66]。对于多基因控制的数量性状,由于无法确定其QTL的准确区域,难以找到与其紧密连锁的分子标记,导致其实际应用价值较低。
QTL所在基因组区域的饱和并在多个环境和遗传背景中进行验证是获得可靠标记的必要前提[69]。因此,应在QTL所在区域进一步设计引物,缩小定位区间。另外,通过蚕豆与豆科模式作物的比较基因组学分析可以有效地验证重要性状相关QTL位点,确定可能的候选基因[70-72],筛选用于后代单株选择的分子标记。同时,开发用于目标性状选择的分子标记,以在育种早期对含有目标性状的个体进行快速、准确的选择,加快育种进程。
参考文献:
[1] 叶 茵. 中国蚕豆学 [M]. 北京: 中国农业出版社, 2003.
[2] 刘玉皎. 调结构,转方式,促进青海蚕豆产业转型升级 [J]. 青海科技, 2018, 25(1): 35-37.
[3] 周俊玲,张蕙杰. 世界蚕豆生产及贸易形势分析 [J]. 世界农业, 2016(11): 107-111.
[4] 冯贝贝,靳 娟,杨 磊,等. 冬枣 (Ziziphus jujube) 果实重要性状和遗传图谱构建及QTL定位研究进展 [J]. 分子植物育种, 2019, 17(24): 8184-8190.
[5] 姜俊烨. 蚕豆微核心种质构建及SSR遗传连锁图谱加密 [D]. 北京:中国农业科学院, 2014.
[6] COOPER J W, WILSON M H, DERKS M F L, et al. Enhancing faba bean (Vicia faba L.) genome resources [J]. Journal of Experimental Botany, 2017, 68(8): 1941-1953.
[7] 瞿华香,张玉烛,张 岳,等. 分子标记辅助选择育种研究进展 [J]. 作物研究, 2008, 22 (S1): 355-358.
[8] VAN DE VEN W T G, WAUGH R, DUNCAN N, et al. Development of a genetic linkage map in Vicia faba using molecular and biochemical techniques [J]. Aspects of Applied Biology, 1991, 27: 49-54.
[9] SUSO M J, MORENO M T. Genetic control of electrophoretic variation for glutamate oxalacetate transaminase (GOT) in Vicia faba L.[J]. Fabis Newsletter Faba Bean Information Service, 1982, 5: 14.
[10]CABRERA A, CUBERO J I, MARTIN A. Genetic mapping using trisomics in Vicia faba L. [J]. Fabis Newsletter Faba Bean Information Service, 1989, 23: 5-7.
[11]SJKDIN J. Induced translocation in Vicia faba L. [J]. Hereditas, 1971, 68: 1-34.
[12]TORRES A M, WEEDEN N F, MARTIN A. Linkage among isozyme, RFLP and RAPD markers in Vicia faba [J]. Theoretical and Applied Genetics, 1993, 85(8): 937-945.
[13]SATOVIC Z, TORRES A M, CUBERO J I. Genetic mapping of new morphological, isozyme and RAPD markers in Vicia faba L. using trisomics [J]. Theoretical and Applied Genetics, 1996, 93: 1130-1138.
[14]PATTO M C V, TORRES A M, KOBLIZKOVA A, et al. Development of a genetic composite map of Vicia faba using F2 populations derived from trisomics plants [J]. Theoretical and Applied Genetics, 1999, 98: 736-743.
[15]ROMN B, TORRES A M, RUBIALES D, et al. Mapping of quantitative trait loci controlling broomrape (Orobanche crenata Forsk.) resistance in faba bean (Vicia faba L.) [J]. Genome, 2002, 45(6): 1057-1063.
[16]AVILA C M, ATOVIC Z, SILLERO J C, et al. Isolate and organ-specific QTLs for ascochyta blight resistance in faba bean [J]. Theoretical and Applied Genetics, 2004, 108: 1071-1078.
[17]ELLWOOD S R, PHAN H T, JORDAN M, et al. Construction of a comparative genetic map in faba bean (Vicia faba L.); conservation of genome structure with Lens culinaris [J]. BMC Genomics, 2008, 9: 380.
[18]DAZ-RUIZ R, SATOVIC Z, AVILA C M, et al. Conrmation of QTLs controlling Ascochyta fabae resistance in different generations of faba bean (Vicia faba L.) [J]. Crop and Pasture Science, 2009, 60: 353-361.
[19]ROMN B, SATOVIC Z, POZARKOVA D, et al. Development of a composite map in Vicia faba, breeding applications and future prospects [J]. Theoretical and Applied Genetics, 2004, 108: 1079-1088.
[20]POARKOVA D, KOBLIKOVA A, ROMN B, et al. Development and characterization of microsatellite markers from chromosome 1-specific DNA libraries of Vicia faba [J]. Biologia Plantarum, 2002, 45: 337-345.
[21]AVILA C M, ATOVIC Z, SILLERO J C, et al. QTL detection for agronomic traits in faba bean (Vicia faba L.) [J]. Agriculturae Conspectus Scientificus, 2005, 70 (3): 65-73.
[22]CRUZ-IZQUIERDO S, AVILA C M, SATOVIC Z, et al. Comparative genomics to bridge Vicia faba with model and closely-related legume species: stability of QTLs for flowering and yield-related traits [J]. Theoretical and Applied Genetics, 2012, 125: 1767-1782.
[23]GUTIRREZ N, PALOMINO C, SATOVIC Z, et al. QTLs for Orobanche spp. resistance in faba bean: identification and validation across different environments [J]. Molecular Breeding, 2013, 32(4): 909-922.
[24]MA Y, BAO S Y, YANG T, et al. Genetic linkage map of Chinese native variety faba bean (Vicia faba L.) based on simple sequence repeat markers [J]. Plant Breeding, 2013, 132 (4): 397- 400.
[25]YANG T, JIANG J Y, ZHANG H Y, et al. Density enhancement of a faba bean genetic linkage map (Vicia faba) based on simple sequence repeats markers [J]. Plant Breeding, 2019, 138 (2): 207-215.
[26]KAUR S, KIMBER R B E, COGAN N O I, et al. SNP discovery and high-density genetic mapping in faba bean (Vicia faba L.) permits identication of QTLs for ascochyta blight resistance [J]. Plant Science, 2014, 217: 47-55.
[27]SALLAM A, ARBAOUI M, ELESAWI M A, et al. Identification and verification of QTL associated with frost tolerance using linkage mapping and GWAS in winter faba bean [J]. Frontiers in Plant Science, 2016, 7: 1098.
[28]WEBB A, COTTAGE A, WOOD T, et al. A SNP based consensus genetic map for synteny-based trait targeting in faba bean (Vicia faba L.) [J]. Plant Biotechnology Journal, 2016, 14: 177-185.
[29]SUDHEESH S, KIMBER R B E, BRAICH S, et al. Construction of an integrated genetic linkage map and detection of quantitative trait loci for ascochyta blight resistance in faba bean (Vicia faba L.) [J]. Euphytica, 2019, 215 (3): 42.
[30]CARRILLO-PERDOMO E, VIDAL A, KREPLAK J, et al. Development of new genetic resources for faba bean (Vicia faba L.) breeding through the discovery of gene-based SNP markers and the construction of a high-density consesus map [J]. Scientific Reports, 2020, 10: 6790.
[31]ARBAOUI M, LINK W, SATOVIC Z, et al. Quantitative trait loci of frost tolerance and physiologically related trait in faba bean (Vicia faba L.) [J]. Euphytica, 2008, 164: 93-104.
[32]DAZ-RUIZ R, TORRES A M, SATOVIC Z, et al. Validation of QTLs for Orobanche crenata resistance in faba bean (Vicia faba L.) across environments and generations [J]. Theoretical and Applied Genetics, 2010, 120: 909-919.
[33]AVILA C M, RUIZ-RODRGUEZ M D, CRUZ-IZQUIERDO S, et al. Identification of plant architecture and yield-related QTL in Vicia faba L. [J]. Molecular Breeding, 2017, 37:88.
[34]SATOVIC Z, AVILA C M, CRUZ-IZQUIERDO S, et al. A reference consensus genetic map for molecular markers and economically important traits in faba bean (Vicia faba L.) [J]. BMC Genomics, 2013, 14(1): 932.
[35]CATT S C, BRAICH S, KAUR S, et al. QTL detection for flowering time in faba bean and the responses to ambient temperature and photoperiod [J]. Euphytica, 2017, 213: 125.
[36]OCAA-MORAL S, GUTIRREZ N, TORRES A M, et al. Saturation mapping of regions determining resistance to ascochyta blight and broomrape in faba bean using transcriptome-based SNP genotyping [J]. Theoretical and Applied Genetics, 2017, 130: 2271-2282.
[37]田瑩莹,侯万伟,刘玉皎. 蚕豆粒型性状的遗传分析及QTL检测 [J]. 分子植物育种, 2018, 16 (4): 1174-1183.
[38]杨生华,刘 荣,杨 涛,等. 蚕豆种质资源种子表型性状精准评价[J]. 中国蔬菜, 2016 (10): 32-40.
[39]沙伟超. 蚕豆子叶颜色性状的SSR标记研究 [D]. 西宁:青海大学, 2017.
[40]AVILA C M, NADAL S, MORENO M T, et al. Development of a simple PCR-based marker for the determination of growth habit in Vicia faba L. using a candidate gene approach [J]. Molecular Breeding, 2006, 17: 185-190.
[41]AVILA C M, ATIENZA S G, MORENO M T, et al. Development of a new diagnostic marker for growth habit selection in faba bean (Vicia faba L.) breeding [J]. Theoretical and Applied Genetics, 2007, 115: 1075-1082.
[42]彭 葵,李锦鸿,李育军,等. 蚕豆的营养与加工研究 [J]. 长江蔬菜, 2019(12): 42-45.
[43]刘珍珍,陈友霞,杨文艺,等. 未成熟蚕豆蛋白组分的分析 [J]. 食品研究与开发, 2019, 40(23): 82-86.
[44]MACAS J, DOLEZEL J, LUCRETTI S, et al. Localization of seed genes on flow-sorted field bean chromosomes [J]. Chromosome Research, 1993, 1: 107-115.
[45]MACAS J, WESCHKE W, BUMLEIN H, et al. Localization of vicilin genes via polymerase chain reaction on microisolated field bean chromosomes [J]. Plant Journal, 1993, 3: 883-886.
[46]TORRES A M, AVILA C M, GUTIERREZ N, et al. Marker-assisted selection in faba bean (Vicia faba L.) [J]. Field Crops Research, 2010, 115: 243-252.
[47]GUTIERREZ N, AVILA C M, RODRIGUEZ-SUAREZ C, et al. Development of SCAR markers linked to a gene controlling absence of tannins in faba bean [J]. Molecular Breed, 2007, 19: 305-314.
[48]GUTIERREZ N, AVILA C M, MORENO M T, et al. Development of SCAR markers linked to zt-2, one of the genes controlling absence of tannins in faba bean [J]. Australian Journal of Agricultural Research, 2008, 59: 62-68.
[49]GUTIERREZ N, AVILA C M, DUC G, et al. CAPs markers to assist selection for low vicine and convicine content in faba bean (Vicia faba L.) [J]. Theoretical and Applied Genetics, 2006, 114: 59-66.
[50]HOU W W, ZHANG X J, YAN Q B, et al. Linkage map of a gene controlling zero tannins (zt-1) in faba bean (Vicia faba L.) with SSR and ISSR markers [J]. Agronomy, 2018, 8: 80.
[51]李仁慧,閆智臣,段廷玉. 蚕豆真菌病害及其研究进展 [J]. 草业科学, 2019, 36(8): 1976-1987.
[52]孙雪梅,易红娟,杨凌峰,等. 多种杀菌剂对蚕豆霜霉病田间防治效果比较 [J]. 农药科学与管理, 2019, 40(7): 60-63.
[53]李 龙,张 芸,郭延平,等. 8种杀菌剂对春蚕豆赤斑病的防治效果 [J]. 植物保护, 2019, 45(3): 245-248.
[54]杜成章,龙珏臣,龚万灼,等. 蚕豆赤斑病抗性的主基因+多基因遗传分析 [J]. 植物保护, 2019, 45(6): 131-137.
[55]王海飞,宗绪晓. 蚕豆种质资源、抗病育种和 QTL 定位及抗逆性研究进展 [J]. 植物遗传资源学报, 2011, 12 (2): 259-270.
[56]ROMN B, SATOVIC Z, AVILA C M, et al. Locating genes associated with Ascochyta fabae resistance in Vicia faba[J]. Australian Journal of Agricultural Research, 2003, 54: 85-90.
[57]SILLERO J C, MORENO M T, RUBIALES D. Characterization of new sources of resistance to Uromyces viciae-fabae in a germplasm collection of Vicia faba [J]. Plant Pathology, 2000, 49: 389-395.
[58]AVILA C M, SILLERO J C, RUBIALES D, et al. Identification of RAPD markers linked to the Uvf-1 gene conferring hypersensitive resistance against rust (Uromyces viciae-fabae) in Vicia faba L. [J]. Theoretical and Applied Genetics, 2003, 107: 353-358.
[59]DAZ-RUIZ R, ROMN B, SATOVIC Z, et al. Validation of QTLs for broomrape resistance in an F6 population of faba bean [C]//AEP. Conference handbook-european conference on grain legumes with international conference on legume genomics & genetics. Dijon, France: European Association for Grain Legume Research, 2004: 122.
[60]DAZ-RUIZ R, SATOVIC Z, ROMN B, et al. QTL analysis of broomrape resistance in faba bean (Vicia faba L.) [C]//KOVACEVIC V, JOVANOVAC S. Proceedings of XL croatian symposium on agriculture. Opatija, Croatia: Faculty of Agriculture, 2005:181-182.
[61]SALEM S, ALGHAMDI H M, MIGDADI M H, et al. Faba bean genomics: current status and future prospects [J]. Euphytica, 2012, 186: 609-624.
[62]REDDY N R, RAGIMASALAWADA M, SABBAVARAPU M M, et al. Detection and validation of stay-green QTL in post-rainy sorghum involving widely adapted cultivar, M35-1 and a popular stay-green genotype B35 [J]. BMC Genomics, 2014, 15 (1): 909.
[63]SALLAM A, DHANAPAL A, LIU S. Association mapping of winter hardiness and yield traits in winter faba bean (Vicia faba L.) [J]. Crop and Pasture Science, 2015, 67: 55-68.
[64]SALLAM A, ARBAOUI M, ELESAWI M A, et al. Identification and verification of QTL associated with frost tolerance using linkage mapping and GWAS in winter faba bean[J]. Frontiers in Plant Science, 2016, 7: 1098.
[65]XIA M Z. Effects of soil drought during the generative development phase of faba bean(Vicia faba)on photosynthetic characters and biomass production[J]. Journal of Agricultural Science, 1994, 122: 67-72.
[66]KHAZAEI H, OSULLIVAN D M, SILLANP M J, et al. Use of synteny to identify candidate genes underlying QTL controlling stomatal traits in faba bean (Vicia faba L.) [J]. Theoretical and Applied Genetics, 2014, 127 (11): 2371-2385.
[67]WANG S C, WONG D, FORREST K, et al. Characterization of polyploidy wheat genomic diversity using a high-density 90 000 single nucleotide polymorphism array [J]. Plant Biotechnology Journal, 2014, 12: 787-796.
[68]王飛飞. 大豆生育期基因TOF7的图位克隆和功能分析[D].长春:中国科学院大学(中国科学院东北地理与农业生态研究所), 2019.
[69]TORRES A M, ROMN B, AVILA C M, et al. Faba bean breeding for resistance against biotic stresses: Towards application of marker technology [J]. Euphytica, 2006, 147 (1/2): 67-80.
[70]GNANASAMBANDAM A, PAULL J G, TORRES A, et al. Impact of molecular technologies on faba bean (Vicia faba L.) breeding strategies[J]. Agronomy, 2012, 2 (3):132-166.
[71]BOREVITZ JO, CHORY J. Genomics tools for QTL analysis and gene discovery [J]. Current Opinion in Plant Biology, 2004, 7: 132-136.
[72]RISPAIL N, KAL P, KISS G B, et al. Model legumes contribute to faba bean breeding [J]. Field Crops Research, 2010, 115 (3):253-269.
(责任编辑:张震林)
收稿日期:2020-07-02
基金项目:国家重点研发计划项目(2019YFD1001300、2019YFD1001304);国家现代农业产业技术体系专项(CARS-08)
作者简介:周仙莉(1995-),女,山东济宁人,硕士研究生,主要从事蚕豆种质资源研究与利用。(E-mail)18797001590@163.com
通讯作者:刘玉皎,(E-mail)13997058356@163.com

