小麦LACS基因家族的全基因组鉴定与表达分析
武雄雄,杨成兰,祁存英,熊辉岩,段瑞君
(1.青海大学生态环境工程学院,青海西宁 810016;2.青海大学农牧学院,青海西宁 810016)
在高等植物中,长链脂酰辅酶A合成酶(LACS)是酰基激活酶(acyl-activating enzyme,AAE)超家族的成员,具有AMP-结合结构域。LACS可活化游离脂肪酸,维持细胞内酰基辅酶A的含量,是植物生长发育必不可少的一类酶[1]。
目前,已经从多种高等植物中分离到LACS基因,其中,拟南芥LACS基因家族有9个基因成员,并且大部分LACS基因在花以及萌发的种子中大量表达,表明这些基因不仅参与花组织的油脂代谢,而且在种子甘油酯的合成中起关键作用[2]。近年来,研究者在棉花[3]、蓖麻[4]、山羊草[5]、水稻[6]、向日葵[7]、牡丹[8]、苹果[9]、甘蓝型油菜[10]等植物中也相继分离到LACS基因家族成员,并对其进行了初步研究。
小麦(TriticumaestivumL.)是一种在全世界广泛种植的农作物之一,我国是小麦主产区之一,随人口的不断扩大,对小麦的需求量也不断增加[11]。研究发现,LACS基因家族在脂质的合成代谢和分解代谢中有着不可代替的作用,对提高植物中脂质产量具有重要意义[12]。但迄今为止,小麦LACS基因家族成员尚未被鉴定。因此,本研究基于小麦全基因组测序数据,利用生物信息学方法对LACS基因家族进行了全基因组鉴定,并进一步对其理化性质、染色体定位、基因结构、保守基序分布、系统进化以及组织和胁迫表达图谱进行分析,以期为后续小麦LACS基因的功能研究提供参考。
1 材料与方法
1.1 小麦LACS基因家族成员的鉴定及其编码蛋白的理化性质分析
用EMBL-Pfam数据库(http://pfam.xfam.org/)下载AMP结构域的隐马尔可夫模型(HMM)文件(AMP-结合结构域:PF00501)[13];用HMMER 3.1软件搜索EnsemblPlants数据库(http://plants.ensembl.org/index.html)下载的小麦基因组文件[14];从拟南芥信息资源(TAIR,http://www.Arabidopsis.org/index.jsp)下载9个拟南芥LACS蛋白[15],从The Rice Annotation Project(RAP,https://rapdb.dna.affrc.go.jp/)下载4个水稻LACS蛋白[16]作为种子序列;用BLASTP搜索小麦LACS蛋白。使用EMBL-Pfam、SMART(http://smart.embl-heidelberg.de/)和 CD Search(Conserved Domain Search Service,https://www.ncbi.nlm.nih.gov/Structure/cdd/wrpsb.cgi)来验证AMP结合结构域,以确定小麦LACS基因家族成员,命名为TaLACS;用ExPASy数据库(https://www.expasy.org/)中对小麦所有LACS蛋白的氨基酸长度、分子量、等电点、不稳定指数进行预测分析。
1.2 TaLACS基因的染色体定位分析
从Ensembl Plants数据库下载小麦的染色体定位数据,用MapChart 2.32软件分析TaLACS基因在染色体上的定位。
1.3 TaLACS蛋白质保守基序和基因结构分析
利用MEME Version 5.1.1(http://meme-suite.org/tools/meme)进行蛋白质保守基序的鉴定。重复次数的为任意次数,最大基序数量为20,以及最佳基序宽度限制在6~200残基之间。使用TBtools软件[17]进行可视化。从Ensembl Plants数据库下载小麦的蛋白和基因结构信息,利用基因结构显示服务器GSDS 2.0(http://gsds.cbi.pku.edu.cn)绘制TaLACS基因结构。
1.4 TaLACS系统发育分析
用小麦、拟南芥和水稻LACS基因家族蛋白序列构建进化树。用Clustal W进行多序列比对,用MEGAX软件构建系统发育树,进化树算法为最大似然法(ML)法,校检参数:重复1 000次。生成的图形使用Evolview v3[18](https://www.evolgenius.info/evolview/#login)进行美化。
1.5 TaLACS基因的组织表达谱分析
从Wheat Exp数据库(http://www.wheat-expression.com/)检索小麦RNA-Seq数据,用Tpm(transcripts per million reads)值评估小麦LACS基因的转录本丰度值。用HEML软件绘制小麦LACS基因表达热图。
1.6 TaLACS基因的启动子分析
从Ensembl Plants数据库下载小麦基因组序列数据,并截取小麦条TaLACS基因起始密码子上游1.5 kb的DNA序列。用Plant CARE软件[19](http://bioinformatics.psb.ugent.be/webtools/plantcare/html/)分析与胁迫响应和植物激素有关的顺式作用元件。
2 结果与分析
2.1 小麦LACS基因家族成员的鉴定结果及其编码蛋白的理化性质
根据拟南芥9个LACS基因和水稻4个LACS基因的蛋白序列,利用HMM和BLASP搜索小麦LACS蛋白序列,之后进行比对,去除冗余序列后合并,利用Pfam和SMART结构域搜索检验LACS候选基因是否含有AMP-bing结构域,最终在小麦基因组中共鉴定出148个LACS候选基因,根据结构域信息以及在基因组的位置,将其命名为TaLACS1~TaLACS148。在ExPASy数据库中对小麦所有LACS蛋白序列进行理化性质预测分析,结果表明,小麦LACS基因家族蛋白氨基酸长度为134~1 593 aa,分子量为 3 654.70~264 379.85 Da,蛋白质等电点为5.23~10.06,均值为7.23,推测大部分蛋白为中性蛋白;不稳定指数中,有48个蛋白的数值大于40,为不稳定蛋白,其余均小于40,为稳定蛋白;总平均疏水指数中,正值的疏水性蛋白和负值的亲水性蛋白各有57和91个;脂溶系数为 78.19~104.94,说明大部分TaLACS蛋白质性质稳定,为亲水脂溶性蛋白。
2.2 TaLACS基因染色体定位分析
染色体物理定位结果(图1)显示,除TaLACS147和TaLACS148两个基因未获得位置信息外,其余146个TaLACS基因不均匀分布在21条染色体上,其中,在5号染色体组上分布最多(36个),1号染色体组上分布最少(9个)。在2D、4A、4B、5A、5B、5D染色体上基因分布比较集中,且有成簇分布现象。
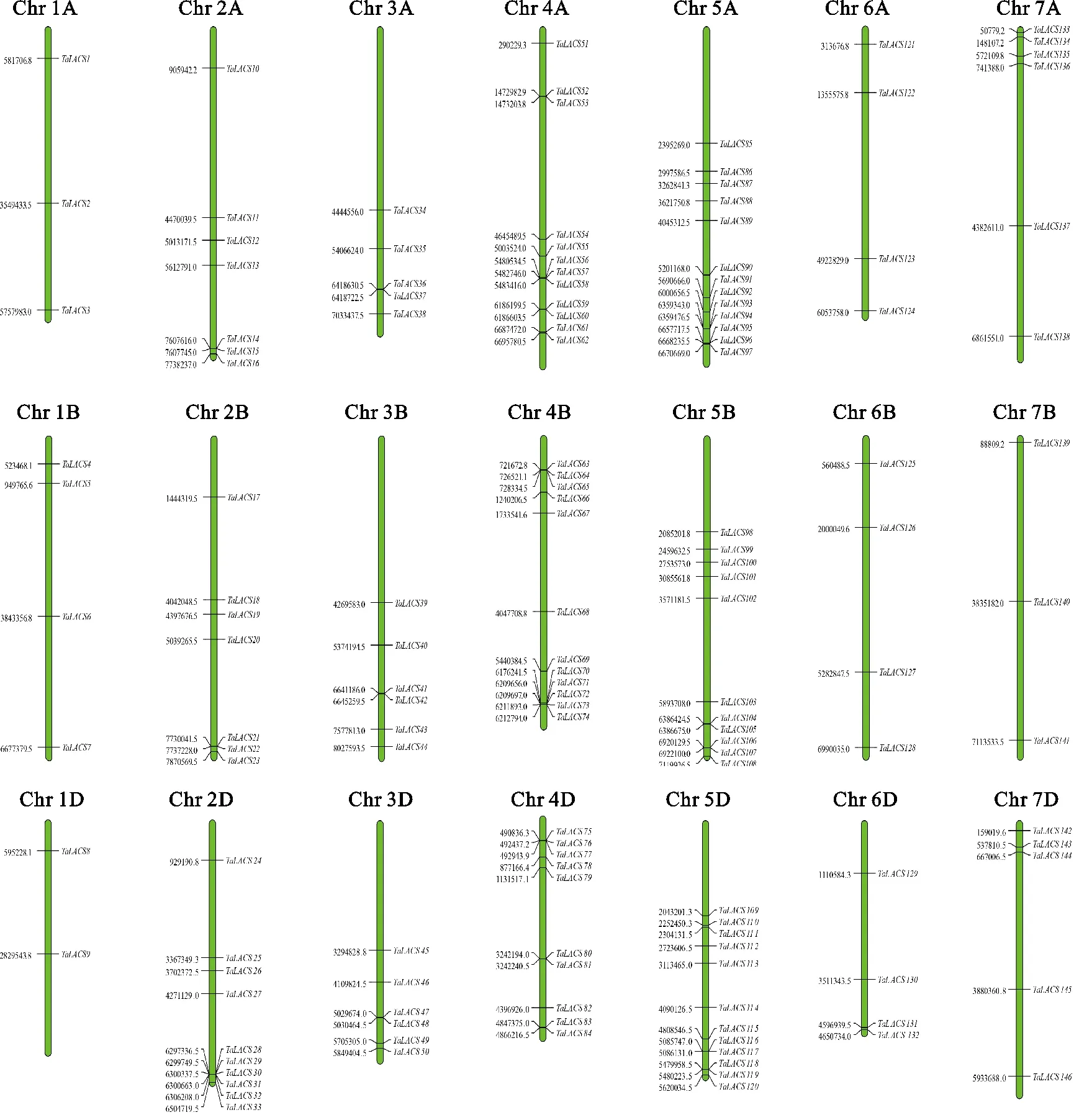
图1 小麦LACS基因在染色体上的定位
2.3 TaLACS蛋白的系统发育分析和氨基酸保守基序和基因结构分析
进化树分析结果(图2a)显示,TaLACS蛋白家族分为6个亚家族,且每个亚家族的保守基序个数与类别基本一致。保守基序分布结果(图2b)显示,TaLACS蛋白保守性较高,共包含5个保守元件,motif1和motif7构成了AMP保守结构域。总的来说,每个TaLACS蛋白的motif为2~15个。TaLACS13、TaLACS20和TaLACS27比较特异,motif长且分散。同时还发现motif1和motif5、motif1和motif16、motif8和motif10经常会一起出现。在基因结构中,大部分小麦TaLACS基因的基因结构基本相似,由编码区和非编码区组成。其外显子子数目为1~23个,内含子数目为0~23个,存在内含子缺失现象。其中,TaLACS3和TaLACS114基因不含内含子。个别TaLACS基因结构只有编码区,无非编区,如TaLACS3、TaLACS19、TaLACS20等25个基因无非编码区,占总基因的16.8%。
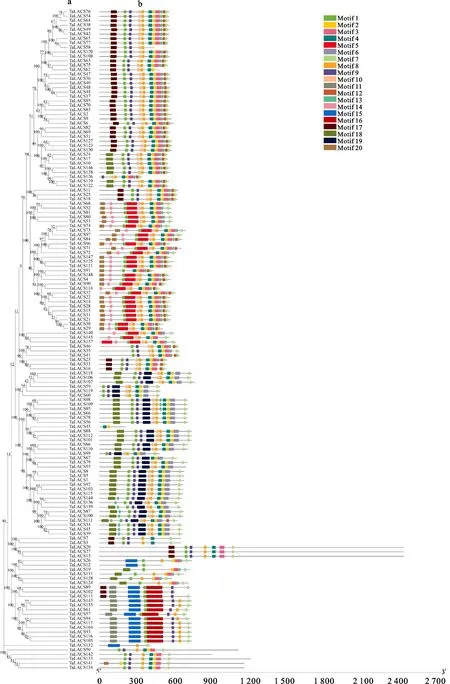
a:小麦LACS蛋白的进化树分析;b:小麦LACS蛋白的保守基序分布。
同时,在148个编码小麦蛋白的TaLACS基因之中检测到55个旁系同源基因对,如TaLACS129的同源基因为TaLACS122,TaLACS79的同源基因为TaLACS5,这些同源基因在TaLACS基因中占到74.3%。为了进一步探究TaLACS基因家族系统发育关系以及该基因家族的进化过程,结合9个拟南芥和4个水稻LACS蛋白序列,利用MEGAX对161个LACS蛋白序列进行系统发育分析。聚类分析结果(图3)表明,148个TaLACS基因家族成员编码的蛋白可分为三大组,分别为GroupI、GroupII和GroupIII。其中,GroupI中有73个成员,GroupII有38个成员,GroupIII有37个成员,GroupIII成员又被细分为GroupIIIa、GroupIIIb、GroupIIIc和GroupIIId 4个亚组,成员数量分别为9、6、6和16。

图3 小麦、拟南芥和水稻LACS蛋白的系统发育树
2.4 TaLACS基因的表达谱分析
利用Wheat Exp数据库中TaLACS基因在种子、叶、根和穗的RNA-Seq数据进行分析,结果(图4a)发现,TaLACS基因的表达模式在不同组织或发育阶段差异明显,总体来说,在营养生长阶段的表达量低于生殖生长阶段。其中,有10个TaLACS基因在种子中表达量较高,有15个TaLACS基因在根中表达量较高,有12个TaLACS基因在叶中表达量较高,有18个TaLACS基因在穗中表达量较高。表明TaLACS基因家族中,在穗和根中高表达的TaLACS基因数目多于在种子和叶中的表达数目。进一步研究发现,TaLACS122和TaLACS129在各个时期和组织中表达量均较高,表明这些基因可能参与调控小麦种子、根、叶和穗的生长发育过程;部分基因在某一组织中表达量较高,而在其他组织中不表达或低表达,如TaLACS1、TaLACS18、TaLACS25等,说明这些基因具有组织特异性表达特点,可能在植物形态建成中起着重要作用。
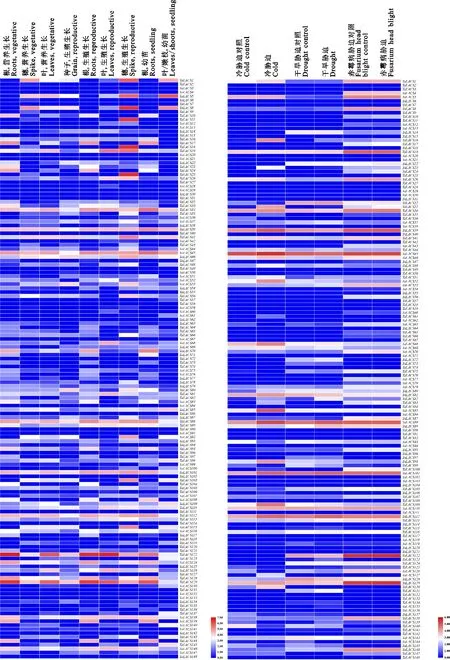
a:TaLACS基因在不同组织中的表达热图;b:TaLACS基因在低温、干旱和赤霉病胁迫下的表达热图。
用Wheat Exp库中TaLACS基因在低温、干旱胁迫和赤霉病诱导的RNA-Seq数据进行分析。结果(图4b)显示,不同胁迫处理后部分TaLACS基因上调表达,响应赤霉病胁迫后TaLACS基因的表达量高于低温和干旱胁迫。其中,TaLACS16、TaLACS23、TaLACS26、TaLACS33、TaLACS39、TaLACS45、TaLACS85、TaLACS88等49个基因在幼苗冷胁迫处理两周后,表达量均明显上升,表明这些基因可能在响应低温胁迫过程中发挥重要作用;在对幼苗进行干旱胁迫后,TaLACS8、TaLACS16、TaLACS51、TaLACS82、TaLACS123、TaLACS139等34个基因上调表达,说明这些基因可能在干旱胁迫响应过程中起关键作用;此外,TaLACS10、TaLACS16、TaLACS24、TaLACS33、TaLACS83、TaLACS139等37个基因在感染赤霉病后上调表达,推测这些基因可能在抗赤霉病感染中起一定作用。
2.5 TaLACS基因的启动子分析
利用Plant CARE软件分析小麦TaLACS基因启动子序列的顺式作用元件,共鉴定到15种与胁迫响应和植物激素有关的顺式作用元件。其中,与胁迫响应相关的元件有5种,分别为厌氧诱导元件(ARE)、真菌引诱应答元件(BOX-W1)、参与防御和应激反应的顺式作用元件(TC-rich repeat)、低温诱导元件(LTR)和干旱诱导响应元件(MBS),其中,含有低温诱导元件和干旱诱导响应元件的TaLACS基因分别占34.9%和 46.0%;与植物激素相关的元件有10种,分别为脱落酸反应元件(ABRE)、生长素响应元件(TGA-element、AuxRR-core)、茉莉酸甲酯响应元件(TGACG-motif、CGTCA-motif)、乙烯应答元件(ERE)、赤霉素响应元件(TATC-box、P-box、GARE-motif)和水杨酸应答元件(TCA-element),其中,含有脱落酸反应元件的TaLACS基因占85.8%。
3 讨 论
近年来,从拟南芥、水稻、苹果、甘蓝型油菜等植物中获得LACS基因[20],并且对其结构和功能作用进行了初步研究,虽然LACS基因在植物蜡质合成和抗胁迫性中发挥着重要作用,但迄今为止,在小麦中还没有对该家族基因进行详细鉴定和功能研究的报道。本研究首次对小麦LACS基因家族进行鉴定分析,依据基因组的注释信息,共获得148个小麦LACS基因,远远多于拟南芥(9个)和苹果(11个)LACS基因家族的成员数目。原因可能是小麦是特殊的六倍体结构,在染色体组A、B、D上存在同源基因[21-22]。Zhang等[9]研究发现,苹果的LACS蛋白长度为596~730 aa,分子量为64.96~79.44 Da,理论等电点为5.56~8.12。Xiao等[10]研究发现,甘蓝型油菜中LACS蛋白长度为129~960 aa,理论等电点为5.11~9.51。本研究发现,TaLACS蛋白长度为134~1 593 aa,分子量为3 654.70~ 264 379.85 Da,理论等电点为5.23~10.06。与苹果和甘蓝型油菜LACS相比,TaLACS蛋白长度较长,分子量较大,且呈中性。蛋白长度和分子量存在差异,推测原因可能是对苹果中LACS蛋白鉴定时只取长度为300~ 1 000 aa的氨基酸序列,而小麦和甘蓝型油菜未作处理;另外小麦较复杂的基因组也可能是原因之一。
将小麦LACS蛋白与拟南芥、水稻LACS蛋白进行系统进化分析,从聚类结果发现,LACS蛋白可分为GroupI、GroupII和GroupIII三大组,其中,GroupIII包含拟南芥和水稻的LACS蛋白,GroupI和GroupII均只有小麦LACS蛋白。GroupIII中TaLACS蛋白有37个,占全部蛋白的四分之一,与AtLACS1和AtLACS2聚在GroupIIIa亚组,与AtLACS3、AtLACS4和AtLACS5聚在GroupIIIb亚组,与AtLACS6和AtLACS7聚在GroupIIIc亚组,与AtLACS8和AtLACS9聚在GroupIIId亚组,这与拟南芥9个LACS蛋白的聚类一致[1];在不同物种中,LACS的分类存在差异。LACS是酰基活化酶(AAE)家族中的成员,根据其聚类结果发现,LACS基因与AAE基因相似性高[23],所以GroupI和GroupII的TaLACS蛋白可能与AAE家族有一定的联系,LACS的进化关系还需要进一步细分。Xu等[24]研究也发现,拟南芥、水稻和小麦LACS基因同时聚类在一个小支上[24],因此,推测TaLACS可能与其他物种LACS基因有相似的功能。Pulsifer等[25]研究发现,AtLACS2和TaLACS115相似性高,AtLACS2可提高脂肪酸含量,推测TaLACS115在小麦中也有类似的功能;Jessen等[26]发现,AtLACS4有形成脂质花粉外壁的功能,与其相似度高的TaLACS100和TaLACS87可能也有类似功能;Fulda等[27]发现,AtLACS6在脂肪酸β-氧化起作用,在种子及幼苗的生长发育阶段中上调表达,推测TaLACS110在小麦种子的萌发过程中也发挥作用。
Fan等[28]研究表明,LACS蛋白可激活长链脂肪酸,然后通过β-氧化作用进行降解、合成细胞脂质,在植物生长发育过程中发挥重要作用。前人研究发现,拟南芥中AtLACS1、AtLACS2和AtLACS4参与蜡的生物合成[29-31],蜡的积累有助于增强植物对ABA和干旱胁迫的耐受性[31-33]。Wang等[34]研究发现,蜡主要在小麦叶片和穗部组织中合成。本研究结果发现,大部分TaLACS基因家族成员在穗和根中上调表达,在叶片和种子中也有上调表达的TaLACS基因,说明大部分TaLACS基因家族成员与蜡合成有关;本研究还发现,TaLACS16、TaLACS39等基因在两种或以上不同胁迫单独处理下均上调表达,其中,TaLACS16在受到冷胁迫、干旱胁迫和赤霉病诱导时均上调表达,响应较为明显。表明小麦TaLACS基因在响应生物胁迫或非生物胁迫中发挥了关键作用,这为LACS基因的功能解析奠定了基础,为进一步研究TaLACS基因在抗生物胁迫和非生物胁迫功能和品种选育提供一定理论指导和实践意义。

