猪源艰难梭菌的分离鉴定与生物学特性分析
张 悦 ,艾伟诚 ,王 斐 ,梁 婉 ,谢思思 ,华 琳 ,李春辉 ,彭 忠 ,吴 斌
(1.华中农业大学动物医学院 农业微生物学国家重点实验室 生猪健康养殖协同创新中心,武汉 430070;2. 湖北省农业科学院畜牧兽医研究所 农业农村部畜禽细菌病防治制剂创制重点实验室,武汉 430070;3.中南大学湘雅医院感染与控制中心,长沙 410008)
艰难梭菌(Clostridioides difficile,也被称作Clostridium difficile)是一种革兰氏阳性、厌氧、产芽孢杆菌,也可感染人和多种动物,引起多种肠道疾病,如腹泻、伪膜性肠炎、巨型结肠等[1]。毒素是艰难梭菌的主要毒力因子,艰难梭菌可分泌产生两种大分子毒素,即毒素A和毒素B,分别由tcdA和tcdB基因编码[2]。此外,在临床上还发现有部分分离株(≤20%)可以编码产生第三种毒素,即二元毒素(binary toxin, CDT)[3]。尽管临床数据显示能编码产生CDT的艰难梭菌引起的感染复发情况较不产生CDT的菌株要严重,然而当前对于CDT在艰难梭菌感染过程中的具体机制依然不清楚[4]。CDT主要由两种基因cdtA和cdtB编码产生[5]。
艰难梭菌感染(Clostridioides difficile infection,CDI)在欧美等发达国家十分严重,仅在美国,每年由于CDI导致的肠炎病例就超过500 000例,造成的经济损失高达4.36~30亿美元[6]。抗生素依然是临床上治疗CDI最有效的方式之一,然而目前被推荐用于治疗CDI的抗生素仅包括甲硝哒唑(Metronidazole, MTZ)、万古霉素(Vancomycin,VAN)和非达霉素(Fidaxomicin, FDX)[7]。其中一个很重要的原因在于艰难梭菌对许多临床上用于治疗细菌感染的抗生素天然具有抗性[8],并且抗生素的使用被认为是导致CDI发生的一个最重要的风险因素[9]。尽管已有研究显示艰难梭菌可以广泛定殖于人和多种动物的肠道中并致病[1],然而当前国内外关于艰难梭菌的研究主要集中在人医领域。尽管目前还没有发现艰难梭菌从人传染给动物的直接证据,然而国外已有研究表明猪源艰难梭菌和人源艰难梭菌之间可能具有共同的来源[10]。因此,开展动物源尤其是食源性动物源艰难梭菌的研究具有十分重要的公共卫生学意义,但相关的研究在国内还几乎处于空白。本研究针对来源于我国部分地区疑似患有肠炎的病猪粪便样品进行艰难梭菌的分离鉴定,并对所分离的菌株进行耐药性测试和全基因组重测序,为了解猪源艰难梭菌的病原学及分子生物学特征奠定基础。
1 材料和方法
1.1 质粒、菌种及试剂 97份疑似患有肠炎的病猪粪便样品收集于湖北省和广东省部分规模化猪场,保存于-20℃备用。人源艰难梭菌LC693(GenBank登录号:NCXL00000000)为一株同时产毒素A、毒素B和二元毒素(A+B+CDT+)的艰难梭菌[11],在本研究中用作对照。胰蛋白大豆琼脂(TSA)、胰蛋白大豆肉汤培养基(TSB)购自美国BD公司;无支原体新生牛血清购自浙江天杭生物科技有限公司。2× Taq Master Mix DNA聚合酶、2× Taq Master Mix Kit购自南京诺唯赞生物科技有限公司;Quick-Load 100 bp DNA Ladder购自美国NEB公司;细菌基因组提取试剂盒购自天根生化科技(北京)有限公司;粪便基因组提取试剂盒购自OMEGA公司;抗生素包括头孢菌素(FOX)、氯霉素(CHL)、万古霉素(VAN)、利福西明(RFX)、克林霉素(CLI)、莫西沙星(MXF)、氨苄西林(AMP)、头孢曲松钠(CRO)、甲硝哒唑(MTZ)、非达霉素(FDX)购自美国Sigma有限公司。
1.2 引物设计 参考文献[12]合成用于检测艰难梭菌16S rDNA基因、毒素A的编码基因tcdA、毒素B的编码基因tcdB以及二元毒素编码基因cdtA和cdtB的5重PCR方法所用的引物(表1),引物由生工生物工程(上海)有限公司合成。

表1 本研究中所使用的引物列表Table 1 Primers used in this study
1.3 粪便样品中艰难梭菌的分离鉴定 按照说明书利用粪便基因组提取试剂盒处理粪便样品并提取粪便基因组DNA,以所提取的DNA为模板,根据文献[12]的报道,利用5重PCR方法对艰难梭菌16Sr DNA基因、毒素编码基因tcdA、tcdB以及cdtA和cdtB进行检测。PCR反应体系(25 μL)为:2×TaqMaster Mix 12.5 μL,引物6.25 μL(终浓度见表1),ddH2O5.25 μL,模板DNA 1 μL。PCR反应程序为:94℃预变性10 min;94℃变性50 s,54℃退火40 s,72℃延伸50s,共35个循环;最后在72℃ 条件下延伸3 min。PCR产物利用3%的琼脂糖进行凝胶电泳分析;利用细菌基因组提取试剂盒提取人源艰难梭菌LC693的基因组作为阳性对照。将艰难梭菌毒素编码基因PCR检测为阳新的粪便样品经75%乙醇溶液处理后,取适量液体在含有10%新生牛血清的TSA平板上划线置于厌氧罐中于37℃培养约48 h,待长出肉眼可见的单菌落,挑取颜色为灰白色,表面粗糙,呈不规则形状,边缘不整齐,并具有特殊气味的菌落进行革兰氏染色鉴定,同时提取细菌的基因组DNA利用上述5重PCR方法作进一步的确认。
1.4 药敏试验 根据美国临床及实验室标准研究所(clinical & laboratory standards institute, CLSI)颁布的《细菌药敏试验规范》(CLSI-M100-S25)中的微量肉汤稀释法(broth microdilution)对所分离的艰难梭菌进行药敏试验;药敏结果参考CLSIM100-S25的规定耐药折点进行判定;对于CLSIM100-S25中未给出判定标准的药物则依据EUCAST或者已发表的文献进行药敏结果判定。
1.5 HuB01的全基因组重测序 将提取的艰难梭菌的基因组DNA送往北京诺禾致源科技股份有限公司进行全基因组重测序。测序平台为Illumina Hiseq Xten(Illumina Inc, San Diego, USA),对得到的下机数据进行过滤处理,去除reads两端低质量碱基(质量值设置为20)及长度过低的reads(默认设为50 bp),同时去除N碱基达到一定比例的reads(默认设为10 bp)以及与Adapter之间overlap超过一定阈值(默认设为15 bp)的reads,最后去除duplication污染后得到有效数据(clean data)。经过数据过滤以及质量控制之后的有效数据利用SPAdes(version 3.9.0)软件进行de novo组装后获取高质量的Contig片段,并进行补洞和优化,最后得到基因组草图序列。利用prokka(version 1.12)软件,按照NCBI原核基因组注释规程(NCBI prokaryotic genome annotation pipeline)的要求对所有基因组序列进行注释。基于NR、PHI、Swiss-Prot、KEGG、COG、GO、Pfam等数据库对编码基因进行功能预测;分别利用VFDB数据库及CADB数据库对基因组中的毒力基因和耐药基因进行预测;此外,由CGView软件完成艰难梭菌全基因组环状图的绘制;将全基因组序列提交至Clostridium difficile MLST database(https://pubmlst.org/cdifficile/)进行序列型(sequence type,ST)的界定。
2 结果
2.1 PCR检测及细菌的分离培养 针对所采集到的97份粪便样品进行艰难梭菌16S rDNA、tcdA、tcdB、cdtA和cdtB的检测,结果发现有80份样品为16S rDNA阳性,其中包括2份样品为tcdA和tcdB阳性;所有粪便样品中均未检测到CDT编码基因cdtA和cdtB。将tcdA和tcdB为阳性的粪便样品经处理后接种含有10%新生牛血清的TSA平板,置于厌氧罐中于37℃培养约48 h,待长出肉眼可见的单菌落,挑取颜色为灰白色,表面粗糙,呈不规则形状,边缘不整齐,并具有特殊气味的菌落进行革兰氏染色鉴定,在油镜下观察可见到蓝紫色的杆菌(图1),说明所分离的细菌为革兰氏阳性细菌。提取所分离菌株的基因组DNA,利用5重PCR进行鉴定,能够检测到艰难梭菌的16S rDNA基因、tcdA和tcdB(图2)。上述结果说明艰难梭菌被成功分离,将所分离的艰难梭菌命名为HuB01。

图1 HuB01的染色结果(×400)Fig.1 Gram staining of HuB01 (×400)

图2 HuB01基因组经5重PCR鉴定的结果Fig.2 The results of HuB01 genomic DNA detected by 5-plex PCR
2.2 药物敏感性测试 用微量肉汤稀释法测定HuB01对头孢菌素、氯霉素、万古霉素、利福西明、克林霉素、莫西沙星、氨苄西林、头孢曲松钠、甲硝哒唑、非达霉素10种抗生素的敏感性,结果显示,HuB01对头孢菌素、氯霉素、克林霉素、氨苄西林、头孢曲松钠耐药,对莫西沙星和万古霉素表现出中度耐药,而对利福西明、甲硝哒唑、非达霉素敏感(表2)。

表2 10种常用 抗生素对HuB01的最小抑菌浓度(MIC)Table 2 Minimum inhibitory concentrations (MICs) of 10 kinds of antibiotics on HuB01
2.3 HuB01全基因组特征 针对HuB01进行全基因组重测序,结果显示HuB01的全基因组大小3 929 112 bp,平均GC含量为28.31%(表3、图3),编码3691个蛋白、63个tRNA和10个rRNA(表3)。3691个基因中含有31个耐药基因(表4)和164个毒力相关基因;将HuB01的全基因组提交至MLST数据库进行分析,结果显示HuB01为ST11。

表3 HuB01的全基因组特征Table 3 Genome characteristics of HuB01
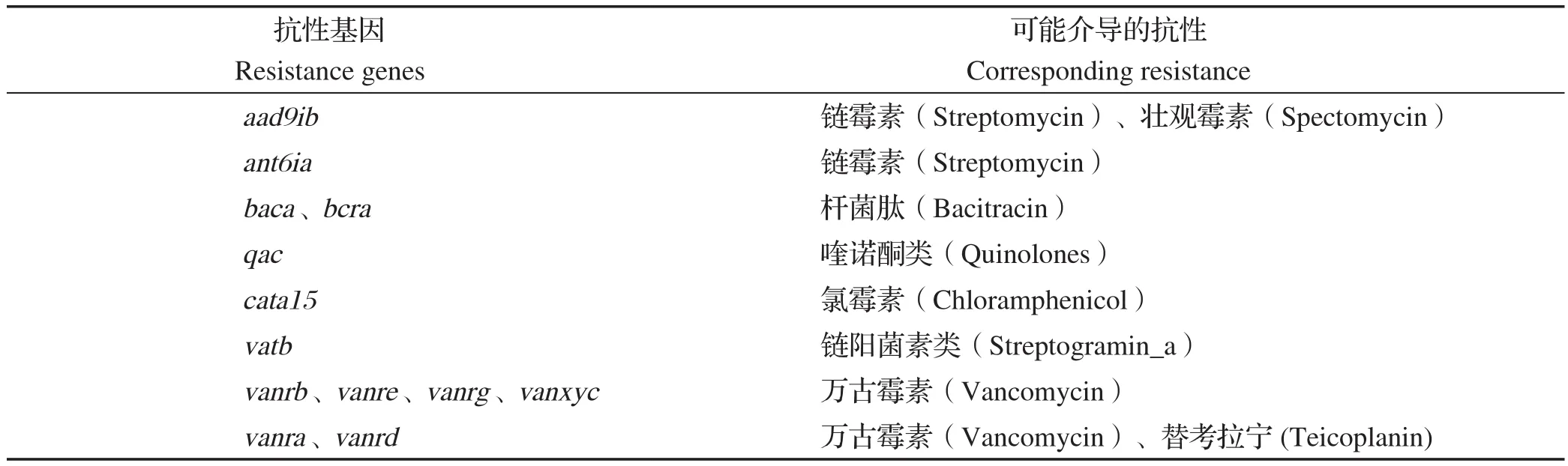
表4 HuB01中所含的耐药基因Table 4 Predicted antimicrobial resistance genes in the genome of HuB01

图4 HuB01基因组圈Fig.4 Circular presentation of the genome of C. diff i cile HuB01
3 讨论
本次研究共采集到97份粪便样品,经5重PCR方法检测,其中80份样品能检测到艰难梭菌的16S rDNA基因,80份样品中有78份样品检测结果为仅16S rDNA阳性,说明在猪群中非产毒艰难梭菌的携带情况比较常见。80份16S rDNA基因为阳性的样品中有两份样品表现为艰难梭菌毒素A和毒素B编码基因呈阳性,说明在猪群中也存在产毒艰难梭菌,这部分菌株应引起重视。下一步将针对所分离的产毒艰难梭菌的致病力进行评估。在本研究中未在猪的粪便样品检出艰难梭菌的二元毒素编码基因。需要指出的是,携带二元毒素的产毒艰难梭菌在人医临床上也较为少见,检出率约为20%[3]。
本研究对从猪场发病猪的粪便中分离的一株产毒艰难梭菌HuB01,药敏试验的结果显示HuB01对头孢菌素、氯霉素、克林霉素、氨苄西林、头孢曲松钠耐药,对莫西沙星和万古霉素表现出中度耐药,对利福昔明、甲硝哒唑、非达霉素敏感,提示猪源艰难梭菌的耐药性十分严重,并呈现多重耐药。在本次研究中艰难梭菌对甲硝哒唑敏感,对万古霉素呈现中度耐药。需要指出的是当前临床上被推荐作为治疗CDI的抗生素仅包括甲硝哒唑、万古霉素和非达霉素[7]。耐万古霉素艰难梭菌的出现将使得治疗CDI的可用抗生素变得更少。HuB01对万古霉素表现出中度耐药不排除和猪场使用万古霉素有关。此外,针对HuB01进行全基因组重测序并进行耐药基因分析发现,在HuB01的基因组中携带有与万古霉素、氯霉素、喹诺酮类等抗性相关的耐药基因(表4),这些基因的存在可能介导了HuB01对相关抗生素的抗性。
本研究所分离鉴定的一株艰难梭菌为ST11型。有报道显示,在猪群中流行的高毒力艰难梭菌RT078的序列型也为ST11[14-15],然而猪源RT078/ST11艰难梭菌产二元毒素,与在北美地区人医临床流行的艰难梭菌高毒力RT027/ST1型菌株具有相似的毒素产生水平[16-17]。有趣的是,本研究中所分离的ST11菌株HuB01不含有二元毒素编码基因。下一步研究将一方面对HuB01进行核糖体分型从而确定其核糖体基因型,另一方面将对HuB01的致病性进行评估,进而更全面地了解HuB01的病原学特征。
综上所述,本研究从来源于湖北和广东部分规模化猪场疑似患有肠炎的病猪粪便样品分离出一株艰难梭菌HuB01,该菌株携带毒素编码基因tcdA和tcdB,不携带二元毒素编码基因cdtA和cdtB;药敏试验显示HuB01对头孢菌素、氯霉素、克林霉素、氨苄西林、头孢曲松钠耐药,对莫西沙星和万古霉素表现出中度耐药,对利福昔明、甲硝哒唑、非达霉素敏感;针对HuB01进行全基因组重测序,结果显示HuB01的全基因组大小为3.93 Mb,平均GC含量为28.31%,基因组中含有多个耐药基因,基于全基因组序列分析发现HuB01为ST11;本研究将为猪源艰难梭菌的病原学及分子生物学特征的研究奠定基础。

