复杂天然产物合成人工生物系统的设计与构建
汪建峰,蒙海林,熊智强,王勇
1中国科学院上海生命科学研究院 植物生理生态研究所 合成生物学重点实验室,上海 200032
2华东理工大学 生物反应器工程国家重点实验室,上海 200237
天然产物一直都是临床药物开发的重要源泉。近十年来基因组、宏基因组测序工作的大规模开展以及新的高效筛选方法的引入,让人们重新认识到自然界中存在着远远超出预期的天然产物或与其合成相关的基因资源,合成生物学技术的出现更是让天然产物的研究获得了全新的机遇[1-2]。合成生物学技术已经深深地影响了天然产物研究的各个环节,天然产物从初期的筛选,到后续的开发、生产和制造正在发生一场深刻的变革。通过挖掘天然产物合成相关的生物学元件,经理性设计并在底盘细胞中集成装配,进一步结合人工生物系统中各功能模块的优化与适配后,可有效地进行珍稀天然产物高效合成[3]。植物源抗疟疾药物青蒿素的中间体青蒿酸的微生物合成即是这一领域最成功的典范。Keasling及其合作者经过十多年的努力,陆续完成了青蒿酸合成途径基因的分离,在酿酒酵母细胞中对青蒿酸合成途径进行重新设计、集成装配、模块适配等工作,最终使酿酒酵母合成了超过25 g/L的青蒿酸[4]。该工作成功地利用微生物发酵大规模生产植物来源的青蒿酸,实现全天候廉价地生产珍稀天然药物。传统天然产物及生物学的研究已经为天然产物合成生物学研究提供了大量的生物学素材,但随着研究的深入,亟需开发更多的生物元件,并对现有的生物元件库进行更新升级和标准化,为构建相关的细胞工厂实现珍稀化合物的大量合成或获得更多的新颖化合物打下基础。此外,随着人工合成系统结构与功能复杂度的增加,也需要进一步发展新的理论和方法,更科学地进行生物元件到功能模块与系统的组装适配[5]。本文将以天然产物合成生物学研究为背景,从生物元件的人工设计,生物元件的挖掘,由生物元件到功能模块系统的组装适配来阐述该领域的研究思路与方法。
1 合成生物学元件的人工理性设计
生物元件 (Biological parts)是具有特定功能的氨基酸或核苷酸序列,如用于基因表达调控的调控元件 (包括启动子、终止子、核糖体结合位点 (RBS)),特定功能的结构元件 (如天然产物合成途径中酶基因)等。它们是生物体最基本的组成单元,也是合成生物学研究中构建人工生命体最基础的砖块[6]。不同来源不同功能的生物元件,可以通过复杂的设计,与其他元件或模块组装成更大规模的具有特定生物学功能的生物回路、装置和系统。因此,生物元件的挖掘与开发是设计与组装更高层次的功能模块和生命系统的基础。传统分子生物学和生物化学研究积累大量的DNA和蛋白质元件,并对许多元件进行了定义。科研人员将这些生物元件整理归档到了一系列开放的元件库中,如美国麻省理工大学的标准生物元件登记库 (Registry of Standard Biological Parts,http://partsregistry.org),Keasling教授课题组开发的The Joint BioEnergy Institute Inventory of Composable Elements(JBEI-ICEs)[7]等。这些元件库的建立大大促进了元件的标准化,方便了其在合成生物学方面的应用。但是相对于浩瀚的天然生物资源,已开发的资源还是极其有限的,仅以微生物资源为例,可培养的微生物仅仅占微生物资源总量的1%,这些未开发的生物资源中蕴藏着大量功能新颖的生物元件[8]。此外更精细复杂的合成生物学系统的设计,对生物元件的功能及生物特性等提出了更高品质的要求。因此设计与开发更高品质的非天然元件,挖掘更多特性新颖的天然元件,是丰富生物元件库的两大策略,也是合成生物学发展最原始的驱动力。根据设计策略的差别主要有两种方法:一是基于随机突变与文库筛选的方法,二是基于模型计算和预测的理性设计方法。
虽然通过构建文库的方法来设计符合预期特性的启动子或RBS等调控元件是一种非常有效的方法,但大规模人工生命系统的构建往往涉及数目庞大的调控元件,而文库的构建与元件的筛选效率低下。此外许多蛋白质元件 (如酶蛋白,调控蛋白等)缺少高通量模型,无法快速地进行文库筛选以获得预期功能的突变体。因此根据掌握的元件序列与功能之间存在的特殊关系,建立计算机模型对元件的关键位点进行改造,理性设计具有预期功能和控制特性的元件是元件设计的重要方向。
De May等[9]利用偏最小二乘 (PLS)回归法建立了大肠杆菌组成型启动子序列与强度关系的预测模型,但预测准确度有待提高。在模型预测的指导下可合理地选择最适强度的启动子并结合启动子敲入 (Knock-in)技术去调控代谢途径上基因的表达,从而实现途径的优化[10]。Rhodius等[11]以大肠杆菌中σE因子识别和结合的启动子为代表,提出了一种基于位置权重矩阵(PWM)模型的强度预测方法。他们把60个σE启动子的−35~+20区域划分为−35、−10、spacer等功能模体,并对每个功能模体进行PWM打分,然后对PWM得分总和与强度值的对数进行线性化拟合,发现部分模体的联合得分与启动子强度具有较好的相关性,R值普遍在0.57至0.77之间。而在RBS设计方面,Salis等[12]通过将描述翻译起始的生物物理模型与最优化算法相结合,提出了一套热力学预测模型用于RBS的设计。它通过预测RBS与核糖体结合的强度,可对蛋白翻译的起始速率及蛋白表达水平进行定量调控。该模型的拟合相关系数较高 (|R|最高达到0.9)。
以上的研究虽然在启动子或RBS强度预测方面已取得了一些进展,但完全基于强度预测的理性设计仍然是十分困难的,主要是由于已有模型预测准确度偏低,难以从真正意义上实现启动子或RBS的序列设计,而且缺乏通用性。实际上,由于序列与强度之间存在十分复杂的非线性对应关系,而现有模型普遍采用线性回归分析方法或经过简单数据处理 (比如取对数)后的回归分析方法难以反映这些复杂的内在关系,从而导致预测准确率偏低。为解决这一难题,本实验室采用了非线性映射能力很强的建模方法——人工神经网络 (Artificial neural network,ANN)来反映这些非线性关系[13],如图1所示,建立了预测效果更佳、普适性更好的预测模型和方法,这些模型和方法可用于指导合成生物学精细调控元件的理性设计。
以原核Trc启动子&RBS调控元件为例,首先基于易错PCR技术及荧光定量筛选法构建了其突变文库 (包含100个序列,相对强度值介于0~3.56之间)。将文库数据随机分为训练集 (不超过90个数据)和测试集 (不少于10个数据)两个集合,用Matlab建立BP神经网络模型,并用训练集数据进行训练。模型对测试集数据的预测误差随着训练集规模的增加而减小,当训练集达到90个数据时,模型的总体预测性能达到最佳。在经过神经元个数、网络层数、训练参数等多种参数综合优化之后,在已构建的1 326 000个模型中,获得了5个预测性能非常好的模型,这些模型解决了BP网络在训练中容易出现的过拟合问题,使模型对测试集数据的预测结果非常接近于实验测量结果。图1给出了最优模型(NET90_19_576)的训练和预测 (测试)结果,其训练和预测的拟合相关系数 (R值)都达到了0.98,远远超过PWM等方法 (图1B),表明人工神经网络模型很好地反映了元件序列和强度之间复杂的非线性关系。
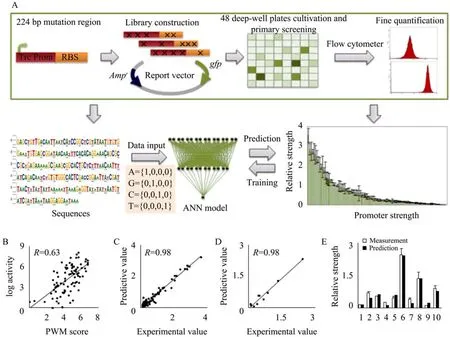
图1 人工神经网络理性设计Trc启动子&RBS调控元件[13](A:调控元件构建的流程图。B:对Trc启动子&RBS元件进行PWM打分后的最佳拟合结果。C:采用ANN方法进行对训练集数据的预测结果与测量结果之间的拟合。D:采用ANN方法进行对测试集数据的预测结果与测量结果之间的拟合。E:人工设计元件的预期值与实验测量值)Fig.1 Rational design of Trc promoter&RBS regulatory element using artificial neural network[13].(A)The workflow of regulatory element library construction.(B)The best fitting results of log Trc promoter&RBS activities with their PWM scores.(C)The predicted relative strengths of promoter&RBS fit with the measured values using the data of training set.(D)The predicted values fit with the measured values using the data of test set.(E)The desired and measured strength of artificially designed elements.
以模型NET90_19_576为基础,进一步对野生型序列的每个碱基进行单位点突变,由模型预测相应的强度值,考察每个位点对强度值的影响,从而得到“关键位点”及“非关键位点”。随后基于“关键位点”的组合突变设计Matlab程序,用NET90_19_576模型实现了全新序列的从头设计。用所设计的元件序列,成功地实现了小肽BmK1在大肠杆菌中有效表达和萜类化合物合成途径的精细定量调控。这不仅有效地解决了小肽蛋白的高效表达及代谢网络的精确调控等问题,也证明了通过人工理性设计的方法所获得的调控序列是可行的、可靠的。该结果表明了人工神经网络方法在精细调控元件理性设计上的应用价值,以及人工设计元件在合成生物学中的巨大应用潜力。
2 天然产物合成相关元件的挖掘、集成与装配
某一特定代谢产物生物合成相关的一组基因,在底盘细胞中重新设计、组装和集成,就能利用底盘细胞已有的代谢网络合成目标产物,实现天然产物的异源高效合成。根据合成途径和构建单元的模块化特性,目前研究人员已经在不同的微生物宿主中 (如大肠杆菌Escherichia coli,酿酒酵母Saccharomyces cerevisiae,橙黄色粘球菌Myxococcus xanthus)[1,14]成功地构建了萜类(单萜,倍半萜,二萜等)[15],聚酮类 (Erythromycin A,Epothilone D 等)[16],苯丙烷酸类 (Resveratrol,Narigenin等)[17],生物碱类化合物[18]的异源合成模块,如表1所示。挖掘和利用已有的生物元件,理性地设计和组装更高效的异源途径是途径重构的努力目标。
2.1 前体合成模块的改造
如何重构底盘细胞,尤其是其前体合成模块,强化前体供应是构建高效底盘细胞的一个重要方面。目前,研究人员重点从前体合成直接相关的途径入手,通过特性元件的替换和表达强度的精细调控,打破途径存在的代谢瓶颈与负调控[19-20]。大量的研究表明引入下游合成模块以后,宿主内源的代谢往往无法为其提供足够的前体和辅因子。如大肠杆菌本身不能合成萜类化合物,仅能由2-甲基-D-赤藻糖醇-4-磷酸 (MEP)途径合成少量的异戊烯基焦磷酸/二甲基烯丙基焦磷酸 (IPP/DMAPP)前体,用于某些必需的细胞结构成分的合成。因此当萜类合成的下游模块引入到野生型大肠杆菌以后,往往只能合成非常微量的目的产物[39]。
甲羟戊酸 (MVA)途径和MEP途径是天然存在的两条萜类化合物前体IPP/DMAPP合成途径。两个模块以不同碳单位 (MVA途径以乙酰-CoA为单位,MEP途径以三磷酸甘油醛和丙酮酸为单位)起始,在合成途径和反应上表现出完全不同的动力学和热力学特性。Keasling等[38]创造性地将酵母MVA途径模块完全引入E.coli中,构建了同时拥有MVA途径和MEP途径两前体合成模块的工程菌株,大大提高了菌株的萜类合成能力。后续的改造发现用来自粪链球菌EnterococcusfaecalisMVA途径模块的HMG-CoA合成酶基因,HMG-CoA还原酶基因代替酵母模块相应的基因后,可以进一步提高异戊烯的合成[40]。这些研究表明不同菌株来源的元件和模块是工程菌株前体合成模块改造的重要资源,通过这些元件和模块的装配能够显著增强底盘细胞前体的供应。虽然外源MVA途径和内源MEP途径模块同时存在的方式,能够显著提高萜类的合成能力,但是我们通过全基因组模型的分析,发现在大肠杆菌中MEP途径比MVA途径具有更大的代谢潜力[41]。随着许多以MEP途径为萜类前体供应途径并含有丰富萜类次级代谢的微生物的陆续报道,MEP途径元件和模块的数据也越来越多,而且不同来源的元件表现出完全不同的催化和调控特性。本研究组选取了链霉菌、芽胞杆菌、糖多胞菌、欧文氏菌等不同菌属为代表,系统地挖掘了相关的dxs、ispD、ispF、idi等MEP途径基因元件 (图2)。结果表明阿维链霉菌来源的dxs2基因,大肠杆菌来源的ispF基因,红霉糖多胞菌来源的ispD基因,枯草芽胞杆菌和阿维来源的idi基因能够明显地强化大肠杆菌MEP途径的代谢通量,增加萜类化合物Amorphadiene和Lycopene的合成。随后我们以过表达阿维链霉菌dxs2基因的工程菌作为出发菌株,通过逐次叠加共表达大肠杆菌ispF基因,红霉糖多胞菌ispD基因,枯草芽胞杆菌idi基因,阿维链霉菌idi基因,获得了共表达阿维链霉菌dxs2和枯草芽胞杆菌idi基因的工程菌,分别使Amorphadiene和Lycopene的产量相对于原始菌株增加了15.5倍和16.5倍。对MEP途径的转录水平进行分析发现,共表达阿维链霉菌dxs2和枯草芽胞杆菌idi基因菌株的内源MEP途径基因的转入水平获得了明显地强化。这些研究结果表明表达某些来源的MEP途径基因不仅能够强化途径中关键步骤的活性,同时也能够激活大肠杆菌内源MEP途径基因的表达,从而有效地解除MEP途径的代谢瓶颈。
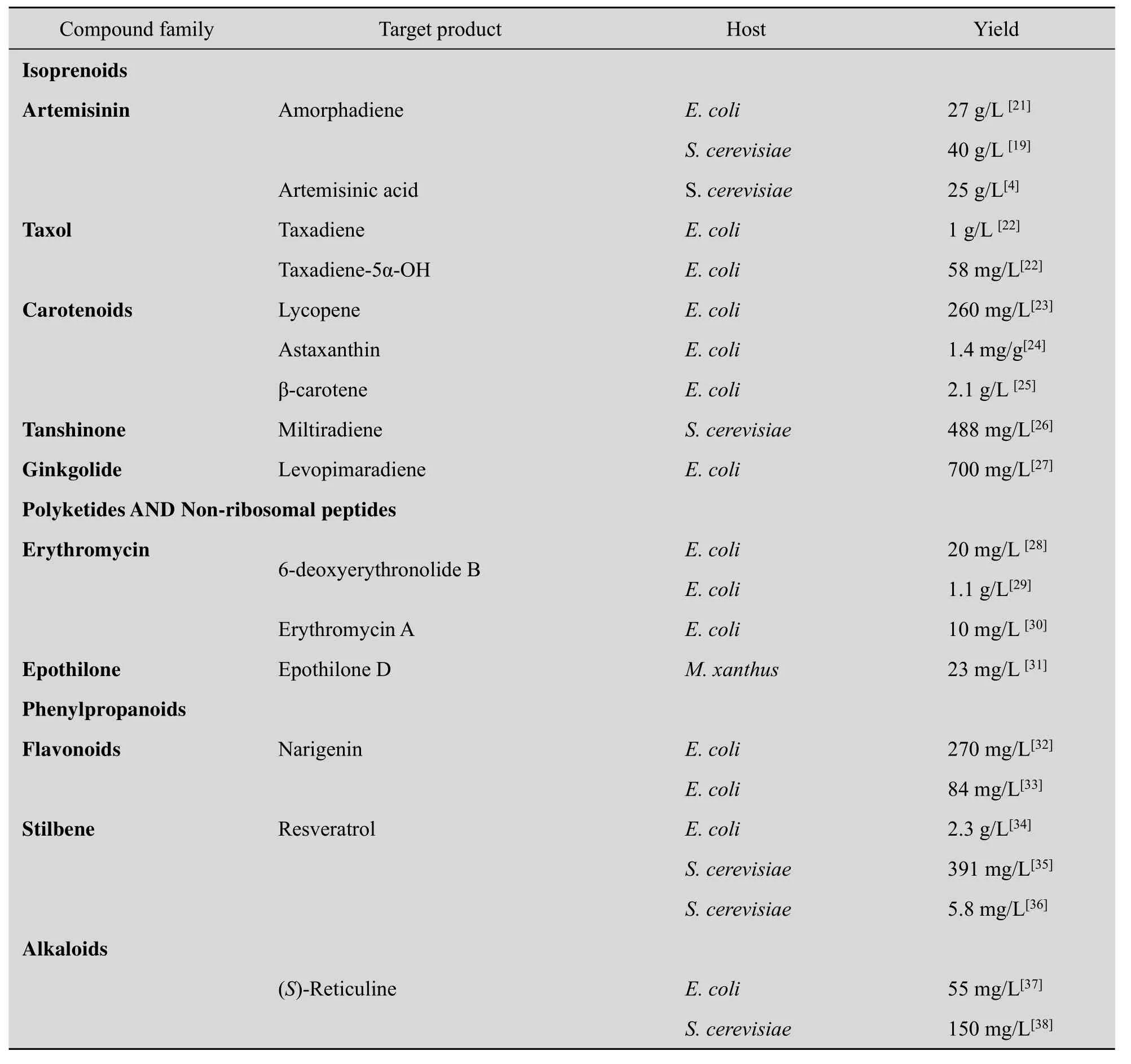
表1 重要天然产物的异源合成Table 1 Heterologous biosynthesis of important natural products
2.2 下游合成模块的设计与装配
随着基因组学研究的深入,越来越多的化合物生物合成途径及其相关基因被陆续阐明。从这些研究结果中可以发现某一个特定化合物的合成途径往往存在于来自不同种属的生物体中,如胡萝卜素类化合物的合成途径广泛分布于植物、细菌、真菌中[42]。同一化合物不同种属来源的途径也表现出明显的差异性,如Lycopene合成途径的八氢番茄红素脱氢酶[43]。此外,同一家族的许多化合物具有相似的母核,共有部分代谢途径。所有这些大自然赋予的生物合成途径多样性和相似性都可以被用于某个化合物异源合成模块的设计和装配。据报道甜叶菊Steviarebaudiana中的甜菊糖和二萜化合物赤霉素的合成途径均以贝壳烯酸作为共有中间代谢物,因此产赤霉素的植物和微生物中与贝壳烯酸合成相关的基因是甜菊糖异源合成途径构建的重要资源[44-45]。本研究组系统地挖掘了数据库中与贝壳烯及终产物甜菊糖类化合物合成相关的基因资源,在E.coli中重构了异源合成途径,实现了由微生物从头合成莱鲍迪苷A(图2)。与所有萜类化合物一样,甜叶菊中莱鲍迪苷A的合成是以IPP和DMAPP为前体,经GGPPS,古巴焦磷酸合酶 (CDPS),贝壳烯合酶 (KS)催化后形成贝壳烯,再经两个P450氧化酶贝壳烯氧化酶 (KO)和贝壳烯酸羟化酶 (KAH)作用形成二萜母核甜菊醇。甜菊醇经四步糖基转移酶反应(UGT85C2,?,UGT74G1,UGT76G1),最终合成莱鲍迪苷A。我们还以更具活性的加拿大红豆杉GGPPS取代甜叶菊的GGPPS,利用小立碗藓Physcomitrella patens的双功能KS[46]取代甜叶菊的CDPS和KS重构贝壳烯合成途径,使贝壳烯的产量获得了明显提高。由于甜菊糖的合成途径中,存在着一步未知的糖基转移酶,因此基于甜叶菊来源的元件目前还无法构建完成的瑞鲍迪苷A合成途径。本课题组利用最大的碳水化合物活性酶 (Carbohydrate-ActiveenZymes,CAZymes)数据库,从1 000多个糖基转移酶中筛选出了真菌斯塔摩酵母Candida bombicola的UGTB1糖基转移酶,可能具有催化甜菊糖合成第二步糖基转移的活性[47]。于是将UGTB1与甜叶菊的KO/P450氧化还原蛋白 (CPR),甜叶菊KAH、UGT85C2糖基转移酶、UGT74G1糖基转移酶、UGT76G1糖基转移酶进一步组装到了甜叶菊合成途径中,最终实现了瑞鲍迪苷A的从头合成。
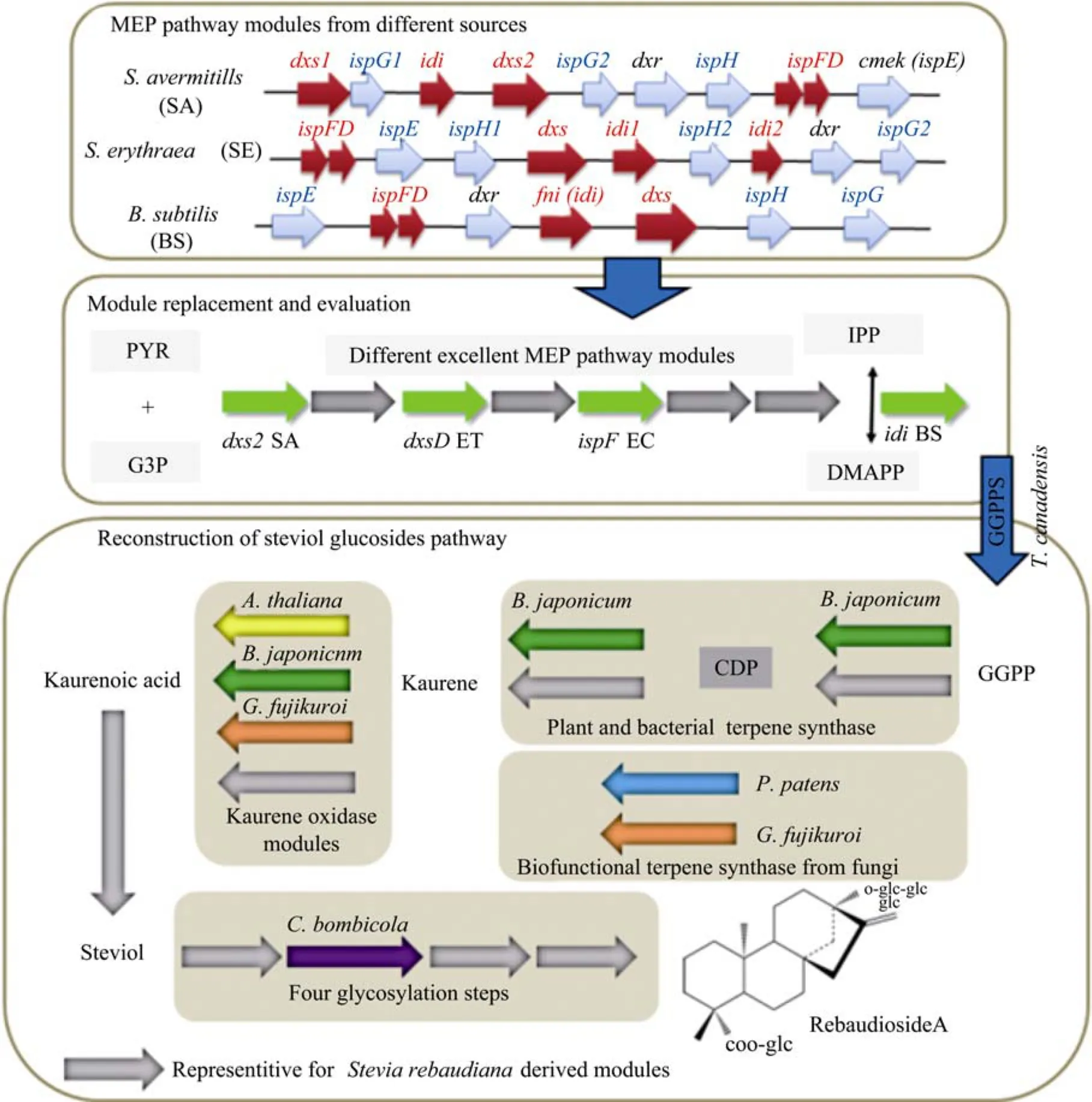
图2 萜类化合物高产菌株的构建.Fig.2 Construction of high-yield isoprenoids producers.
在重构紫杉醇途径的过程中,受到植物来源的元件有限,而且往往受到物种亲缘关系较远的影响,在微生物宿主中表达与活性均不甚理想,需要经过密码子优化及催化特性的改造。考虑到产紫杉醇内生真菌资源的多样性,本课组以Ta x o l生物合成基因簇中紫杉二烯合酶(Taxadiene synthase,TS)、C-13苯丙氨酸侧链辅酶A酰基转移酶 (C-13 phenylpropanoid side chain-CoA acyltransferase,BAPT)和 10巴卡亭III酰基转移酶 (10-deacetylbaccatin III-10-O-acetyltransferase,DBAT)为靶向设计了同源引物,通过对红豆杉来源的内生真菌样品进行保守序列PCR,从产紫杉醇的内生真菌中筛选出了新的TS、BAPT和DBAT功能元件[48]。经简单的序列分析发现这些真菌来源的元件与植物来源的相比,具有很低的同源性,可以为异源Taxol合成途径的组装提供更多元化选择。
2.3 产物转运模块的装配
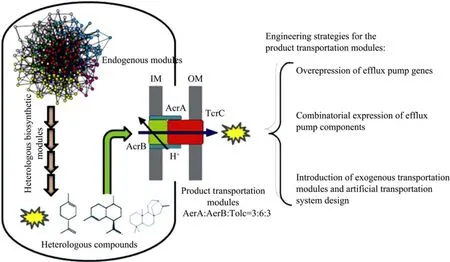
图3 产物转运模块的改造.Fig.3 Modification of the product transportation module.
在底盘细胞中通过重构合成途径,进行天然产物的异源合成,如何解决异源产物的转运,增加底盘细胞对产物的耐受性是一个关键问题,这也是提高工程菌株产物合成能力的重要手段。许多产生复杂次生代谢物的宿主具有多种天然的机制以确保产物可以运出胞外,并使诉诸本身对特定产物具有一定的耐受性,如广泛存在的多药耐药泵系统、热激蛋白的表达、细胞膜的修饰等机制。多药外排泵系统 (Multidrug Efflux Pump)是一类广泛存在的能够将胞内的药物或毒性物质排除胞外的转运系统,如大肠杆菌的三组分耐药泵AcrAB-TolC系统,绿脓假单胞菌Pseudomonas aeruginosa中的MexAB-OprM系统等具有非常宽泛的底物特异性,可以将多种抗生素、有机溶剂、金属离子转运到细胞外[49]。在萜类化合物的异源合成研究中,本课题组以大肠杆菌的三组分耐药泵作为改造对象 (图3),通过逐个强化大肠杆菌内源AcrAB-TolC各组分的表达,发现外膜通道蛋白TolC及外排转运蛋白AcrB的过表达能够显著提高萜类化合物Amorphadiene和贝壳烯的合成[50]。考虑到三组分耐药泵是一个以AcrA:AcrB:TolC=3:6:3精细装配的分子机器,我们对3个组分进行两两组合强化表达,同时进行各组分比例的精细调控。最终组合TolC&TolC&AcrB使Amorphadiene的比产率提高了118%,而AcrA&TolC&AcrB使贝壳烯的比产率提高了104%。这些结果说明强化工程菌株的转运系统确实能够有效地提高目标产物的合成。三组分耐药泵作为最主要的转运系统,针对其的改造具有非常重要的意义。除大肠杆菌内源的转运系统,许多耐药菌也存在着类似的系统。于是我们在大肠杆菌中过表达了来自绿脓假单胞菌的MexAB-OprM转运系统,当单独表达外排转运蛋白MexB时,Amorphadiene和贝壳烯的比产率提高了70%,比单独表达TolC或AcrB取得了更明显的效果。此外我们也尝试异源转运系统的组合表达及异源内源组分的共表达,相比于前述AcrAB-TolC组分的组合表达,目标产物产率没有能够取得更进一步的提高,但是异源耐药泵仍旧是转运系统重构的重要元件。
3 合成生物学系统的优化与适配
异源合成模块的导入使原有宿主系统获得合成某种目标产物的能力,但其生产效率及产率通常仍处于较低水平,无法满足工业生产的需求。这是由于异源合成模块与原有宿主系统之间尚存在不适配问题。因此,从整体上对宿主系统进行优化并改善其与异源合成模块之间的互作与适配,是大幅提高下游目标产物合成效率的重要研究内容。
3.1 异源合成系统的优化与设计
尽管经典的诱变育种及代谢工程策略已成功应用于优化宿主系统和提高产率,但诱变育种具有很大的随机性,给定向筛选带来很大的困难,费时费力;代谢工程则对多基因决定的性状难以达到理想效果[51],而且难以发现与目标产物代谢不直接相关的关键靶点。近年来,基因组尺度代谢模型 (GSMM)的提出[52]使菌种改造研究提升到系统的高度。基于GSMM,采用通量平衡分析 (FBA)[53]、最小代谢调整分析 (MOMA)[54]等in silico分析方法,可发现某些影响目标产物合成的关键基因靶点,尤其是与产物合成不密切相关的靶点。通过计算机辅助的理性分析设计,可以有效减少实验的盲目性,提高工作效率并降低实验成本。为此,本实验室在FBA、MOMA等经典算法的基础上,分别设计了两套算法(FDCA和LMOMA-Based)[55],并用于预测一些可能提高目标产物产率的关键基因改造靶点(图 4)。
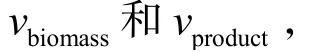

图4 基于FDCA及LMOMA算法的异源合成系统设计与优化Fig.4 Heterologous biosynthetic system design and optimization based on FDCA and LMOMA algorithm.

我们以大肠杆菌为宿主、葡萄糖为底物、红霉素中间体6dEB作目标产物为例,通过FDCA和LMOMA-Based算法获得了一些潜在基因敲除位点[55],为提高6dEB异源合成产率奠定了理论基础。
3.2 元件与模块的适配
基于GSMM的整体代谢优化可改善宿主的网络流量分布,使代谢流量尽可能多地流向目标产物的合成。然而,异源合成模块与宿主系统之间并非是完美协作的,各元件之间、下游与前体模块之间如何实现合理的搭配,消除元件、模块之间因不适配而引起中间代谢物积累与能量的消耗,也是增强目的产物合成的重要手段。这是因为细胞生命活动是时间和空间上都是多层次交互作用 (DNA复制,基因转录,蛋白质翻译,物质与能量代谢,代谢物的运输与分泌,细胞信号的转导等)的结果,仅仅基于不同模块代谢网络之间代谢流量的分析,进行生物模块之间的简单改造是无法实现系统的最佳适配。Kim等[56]通过研究大肠杆菌中一条与磷酸吡哆醛合成相关的新代谢途径与细胞已知代谢网络之间的相互作用发现:当新的或外源途径引入到已有的细胞网络后,存在着多个层次的相互作用:1)新代谢产物与已有代谢网络的干扰;2)已有代谢产物与新代谢网络的干扰;3)新代谢途径中酶的某些非特异性活性,造成某些中间代谢物和能量因子被消耗,同时新代谢产物对细胞已有调控网络的干扰等。异源系统中由于各种来源,不同特性的元件、模块之间的不适配必然会存在类似的负相互作用,因此元件、模块适配和重组系统调试是异源合成研究的新热点。
本实验室在in silico代谢网络分析及优化的基础上,结合转录组芯片分析及基于13C标记的代谢流量分析,可从不同层次上对异源合成模块与宿主系统间的互作及适配机制进行探讨。例如,通过对6dEB在大肠杆菌中的异源合成进行不同层次的考察,发现不同功能模块之间在真实情形下与“理想情形”下 (细胞完全以产物合成为目标)存在较大的表型差异,包括转录水平及代谢水平上的整体差异,反映了不同模块间确实存在适配性问题,并直接影响了目标产物的合成效率。因此,通过对模块内与模块间的相关基因进行改造,缩小真实情形与“理想情形”的差异,改善模块间的适配性问题,是提高目标产物异源合成效率的关键所在,也是本实验室正在开展的工作。
4 展望
随着研究材料的丰富和理论方法与技术的不断完善进步,合成生物学技术将彻底变革传统天然产物开发的研究思路,并最终实现珍稀天然产物的大规模制造和新化合物的发现。更多新天然元件的挖掘将进一步拓展人类认知与合成生物学应用的范围,丰富新合成生物学系统构建的生物砖,同时运用新的元件也能够改进已有系统的效率,发挥系统优化的作用。新的元件装配和系统适配思路与方法的实施,也大大提升人工生命系统的性能与品质。集成运用以上方法后,异源产物的合成效率获得了显著的提升,许多珍贵的化合物产量由初始的毫克/升水平增加到了克/升,甚至几十克/升。部分产品的工艺 (如青蒿酸)即将取代了原有的生产方式,正在进行中试和工业规模的放大研究。因此从生物元件挖掘设计到模块系统的组装适配各方面的思路,方法与技术的革新,将全面促进合成生物学技术发展与成熟。
[1]Wang JF,Xiong ZQ,Meng HL,et al.Synthetic biology triggers new era of antibiotics development.Subcell Biochem,2012,64:95−114.
[2]MitchellW.Naturalproducts from synthetic biology.CurrOpin Chem Biol,2011,15(4):505−515.
[3]Lu ZG,Wang JF,Meng HL,et al.Synthetic biology and natural products development.Chin Bull Life Sci,2011,23(9):900−911(in Chinese).
卢志国,汪建峰,蒙海林,等.合成生物学与天然产物开发.生命科学,2011,23(9):900−911.
[4]Paddon CJ,Westfall PJ,Pitera DJ,et al.High-level semi-synthetic production of the potent antimalarial artemisinin.Nature,2013,496:528−532.
[5]Purnick PE,Weiss R.The second wave of synthetic biology:from modules to systems.Nat Rev Mol Cell Biol,2009,10(6):410−422.
[6]Canton B,Labno A,Endy D.Refinement and standardization of synthetic biological parts and devices.Nat Biotechnol,2008,26(7):787−793.
[7]Ham TS,Dmytriv Z,Plahar H,et al.Design,implementation and practice of JBEI-ICE:an open source biological part registry platform and tools.Nucleic Acids Res,2012,40(18):e141.
[8]Xiong ZQ,Wang JF,Hao YY,et al.Recent advances in the discovery and development of marine microbial natural products.Mar Drugs,2013,11(3):700−717.
[9]De Mey M,MaertensJ,Lequeux G,etal.Construction and model-based analysis of a promoter library forE.coli:an indispensable tool for metabolic engineering.BMC Biotechnol,2007,7(1):34.
[10]De Mey M,Maertens J,Boogmans S,et al.Promoter knock-in:a novel rational method for the fine tuning of genes.BMC Biotechnol,2010,10(1):26.
[11]Rhodius VA,Mutalik VK.Predicting strength and function for promoters of theEscherichia colialternative sigma factor,sigmaE.Proc Natl Acad Sci USA,2010,107(7):2854−2859.
[12]Salis HM,Mirsky EA,Voigt CA.Automated design of synthetic ribosome binding sites to control protein expression.Nat Biotechnol,2009,27(10):946−950.
[13]Meng HL,Wang JF,Xiong ZQ,et al.Quantitative design of regulatory elements based on high-Precision strength prediction using artificial neural network.PLoS ONE,2013,8(4):e60288.
[14]Zhang H,Wang Y,Pfeifer BA.Bacterial hosts for natural product production.Mol Pharm,2008,5(2):212−225.
[15]Misawa N.Pathway engineering for functional isoprenoids.Curr Opin Biotechnol,2011,22(5):627−633.
[16]Gao X,Wang P,Tang Y.Engineered polyketide biosynthesis and biocatalysis inEscherichia coli.Appl Microbiol Biotechnol, 2010, 88(6):1233−1242.
[17]Wang Y,Chen S,Yu O.Metabolic engineering of flavonoids in plants and microorganisms.Appl Microbiol Biotechnol,2011,91(4):949−956.
[18]Chow YL,Sato F.Metabolic engineering and synthetic biology for the production of isoquinoline alkaloids//Chandra S,Lata H,Varma A,Eds.Biotechnology for Medicinal Plants. Berlin,Heidelberg:Springer,2013:327−343.
[19]Westfall PJ,Pitera DJ,Lenihan JR,et al.Production of amorphadiene in yeast,and its conversion to dihydroartemisinic acid,precursor to the antimalarial agent artemisinin.Proc Natl Acad Sci USA,2012,109:E111-E118.
[20]Martin VJ,Pitera DJ,Withers ST,et al.Engineering a mevalonate pathway inEscherichia colifor production of terpenoids.Nat Biotechnol,2003,21(7):796−802.
[21]Tsuruta H,Paddon CJ,Eng D,et al.High-level production of amorpha-4,11-diene,a precursor of the antimalarial agent artemisinin,inEscherichia coli.PLoS ONE,2009,4(2):e4489.
[22]Ajikumar PK,Xiao WH,Tyo KEJ,et al.Isoprenoid pathway optimization for taxol precursor overproduction inEscherichia coli.Science,2010,330(6000):70−74.
[23]Kim SW,Kim JB,Ryu JM,et al.High-level production of lycopene in metabolically engineeredE.coli.Process Biochem,2009,44(8):899−905.
[24]Lemuth K,Steuer K,Albermann C.Engineering of a plasmid-freeEscherichia colistrain for improvedin vivobiosynthesis of astaxanthin.Micorob Cell Fact,2011,10(1):29.
[25]Zhao J,Li Q,Sun T,et al.Engineering central metabolic modules ofEscherichiacolifor improving β-carotene production.Metab Eng,2013,17(0):42−50.
[26]Dai Z,Liu Y,Huang L,et al.Production of miltiradiene by metabolically engineeredSaccharomycescerevisiae. Biotechnol Bioeng,2012,109(11):2845−2853.
[27]Leonard E,AjikumarPK,ThayerK,etal.Combining metabolic and protein engineering of a terpenoid biosynthetic pathway for overproduction and selectivity control.Proc Natl Acad Sci USA,2010,107(31):13654−13659.
[28]PfeiferBA,AdmiraalSJ,Gramajo H,etal.Biosynthesis of complex polyketides in a metabolically engineered strain ofE.coli.Science,2001,291(5509):1790−1792.
[29]Lau J,Tran C,Licari P,et al.Development of a high cell-density fed-batch bioprocess forthe heterologous production of 6-deoxyerythronolide B inEscherichia coli.J Biotechnol,2004,110(1):95−103.
[30]Zhang H,Wang Y,Wu J,etal.Complete biosynthesis oferythromycin A and designed analogs usingE.colias a heterologous host.Chem Biol,2010,17(11):1232−1240.
[31]Lau J,Frykman S,Regentin R,et al.Optimizing the heterologous production of epothilone D inMyxococcus xanthus.Biotechnol Bioeng,2002,78(3):280−288.
[32]Fowler ZL,Gikandi WW,Koffas MA.Increased malonyl coenzyme A biosynthesis by tuning theEscherichiacolimetabolic network and its application to flavanone production.Appl Environ Microbiol,2009,75(18):5831−5839.
[33]Santos CNS, Koffas M, Stephanopoulos G.Optimization of a heterologous pathway for the production of flavonoids from glucose.Metab Eng,2011,13:392−400.
[34]Lim CG,Fowler ZL,Hueller T,et al.High-yield resveratrol production in engineeredEscherichia coli.Appl Environ Microbiol,2011,77(10):3451−3460.
[35]Sydor T,Schaffer S,Boles E.Considerable increase in resveratrol production by recombinant industrial yeast strains with use of rich medium.Appl Environ Microbiol, 2010, 76(10):3361−3363.
[36]Shin SY,Han NS,Park YC,et al.Production of resveratrol from p-coumaric acid in recombinantSaccharomyces cerevisiaeexpressing 4-coumarate:coenzyme A ligase and stilbene synthase genes.Enzyme Microb Technol,2011,48(1):48−53.
[37]Minami H,Kim J-S,Ikezawa N,et al.Microbial production of plant benzylisoquinoline alkaloids.Proc Natl Acad Sci USA,2008.
[38]Hawkins KM, Smolke CD. Production of benzylisoquinoline alkaloids inSaccharomyces cerevisiae.Nat Chem Biol,2008,4(9):564−573.
[39]Yuan LZ,Rouviere PE,Larossa RA,etal.Chromosomal promoter replacement of the isoprenoid pathway for enhancing carotenoid production inE.coli.Metab Eng,2006,8(1):79−90.
[40]Yang J,Xian M,Su S,et al.Enhancing production of bio-isoprene using hybrid MVA pathway and isoprene synthase inE.coli.PLoS ONE,2012,7(4):e33509.
[41]Meng H,Wang Y,Hua Q,et al.In silicoanalysis and experimental improvement of taxadiene heterologous biosynthesis inEscherichiacoli.Biotechnol Bioprocess Eng,2011,16(2):205−215.
[42]Paniagua-MichelJ,Olmos-Soto J,Ruiz MA.Pathways of carotenoid biosynthesis in bacteria and microalgae.Methods Mol Biol,2012,892:1−12.
[43]Schaub P,Yu Q,Gemmecker S,et al.On the structure and function of the phytoene desaturase CRTI fromPantoeaananatis,a membraneperipheral and FAD-dependent oxidase/isomerase.PLoS ONE,2012,7(6):e39550.
[44]Hedden P,Thomas SG.Gibberellin biosynthesis and its regulation.Biochem J,2012,444(1):11−25.
[45]Brandle JE, Telmer PG. Steviol glycoside biosynthesis. Phytochemistry, 2007, 68(14):1855−1863.
[46]HayashiK,Kawaide H,NotomiM,etal.Identification and functional analysis of bifunctional ent-kaurene synthase from the mossPhyscomitrella patens.FEBS Lett,2006,580(26):6175−6181.
[47]Van Bogaert INA,Zhang J,Soetaert W.Microbial synthesis of sophorolipids.Process Biochem,2011,46(4):821−833.
[48]Xiong ZQ,Yang YY,Zhao N,et al.Diversity of endophytic fungi and screening of fungal paclitaxel producer from Anglojap yew,Taxus xmedia.BMC Microbiol,2013,13(1):71.
[49]Webber MA,Piddock LJV.The importance of efflux pumps in bacterial antibiotic resistance.J Antimicrob Chemother,2003,51(1):9−11.
[50]Wang JF,Xiong ZQ,Li SY,et al.Enhancing isoprenoid production through systematically assembling and modulating efflux pumps inEscherichia coli.Appl Microbiol Biotechnol,2013,DOI:10.1007/s00253−013−5062−z.
[51]Zhao XQ,Bai FW,Li Y.Application of systems biology and synthetic biology in strain improvement for biofuel production.Chin J Biotech,2010,26(7):880−887(in Chinese).
赵心清,白凤武,李寅.系统生物学和合成生物学研究在生物燃料生产菌株改造中的应用.生物工程学报,2010,26(7):880−887.
[52]Edwards JS,Palsson BO.TheEscherichia coliMG1655insilicometabolic genotype: its definition,characteristics,and capabilities.Proc Natl Acad Sci USA,2000,97(10):5528−5533.
[53]Edwards JS,Palsson BO.Metabolic flux balance analysis and thein silicoanalysis ofEscherichia coliK-12 gene deletions.BMC Bioinformatics,2000,1(1):1−1.
[54]Segrè D,Vitkup D,Church GM.Analysis of optimality in naturaland perturbed metabolic networks.Proc Natl Acad Sci USA,2002,99(23):15112−15117.
[55]Meng HL,Lu ZG,Wang Y,et al.In silicoimprovement of heterologous biosynthesis of erythromycin precursor 6-deoxyerythronolide B inEscherichia coli.Biotechnol Bioprocess Eng,2011,16(3):445−456.
[56]Kim J,Copley SD.Inhibitory cross-talk upon introduction of a new metabolic pathway into an existing metabolic network.Proc Natl Acad Sci USA,2012,109(42):E2856−2864.

