雷蒙德氏棉HSP70基因家族的进化分析及其同源基因在陆地棉中的表达分析
张毓婷,王敏华,陈家栋,戎均康,3,丁明全,3
1. 浙江农林大学,亚热带森林培育国家重点实验室培育基地,临安 311300;
2. 浙江农林大学农业与食品科学学院,浙江省农产品品质改良技术研究重点实验室,临安 311300;
3. 中国农业科学院棉花研究所,棉花生物学国家重点实验室,安阳 45500
雷蒙德氏棉HSP70基因家族的进化分析及其同源基因在陆地棉中的表达分析
张毓婷1,2,王敏华1,2,陈家栋2,戎均康2,3,丁明全2,3
1. 浙江农林大学,亚热带森林培育国家重点实验室培育基地,临安 311300;
2. 浙江农林大学农业与食品科学学院,浙江省农产品品质改良技术研究重点实验室,临安 311300;
3. 中国农业科学院棉花研究所,棉花生物学国家重点实验室,安阳 45500
热激蛋白70家族(HSP70)是一类在植物中高度保守的分子伴侣蛋白,在细胞中协助蛋白质正确折叠。文章利用隐马可链夫模型(HMM)在雷蒙德氏棉(Gossypium raimondii L.)全基因组范围内进行HSP70基因家族成员进化分析,共得到30个HSP70家族成员。利用生物信息学对雷蒙德氏棉HSP70基因的结构、染色体分布、基因倍增模式以及系统进化进行分析,结果表明,HSP70基因家族根据亚细胞定位结果可分为不同的基因亚家族,各亚家族中HSP70基因具有相对保守的基因结构;染色体片段重复和串联重复是雷蒙德氏棉HSP70基因家族扩增的主要方式。通过对不同物种的HSP70基因家族进行系统进化分析可知,HSP70亚组的分化发生在单细胞植物形成前,且细胞质型 HSP70成员大量扩增。比较陆地棉棉纤维发育不同时期的深度测序表达谱,发现HSP70基因可能参与棉纤维的生长发育。本研究结果有助于了解棉属植物 HSP70基因家族的功能,以期为深入研究棉纤维发育过程中的分子调控机理提供基础。
棉纤维;HSP70家族;深度测序;基因结构;系统进化分析
棉纤维是全球重要的纺织原料之一,具有极其重要的经济价值。棉纤维是单细胞发育的典型模型,是研究细胞生长发育的极佳材料,了解其发育过程的机理具有重大意义[1]。棉纤维由胚珠外珠被表皮层的单细胞分化伸长形成,需要经历纤维原始细胞的分化和突起、初生壁伸长、次生壁增厚和脱水成熟4个依次并相互重叠的过程,一般历时45~50 d,涉及上千个基因的特异表达和蛋白相互作用[2,3]。目前,陆地棉(Gossypium hirsutum)是栽培面积及经济价值最大的栽培棉种,异源四倍体(AD染色体组)的形成约发生在1~2百万年前,普遍认为其D组染色体组的供体为雷蒙德氏棉(G. raimondii)。二倍体棉花雷蒙德氏棉基因组的测序工作已完成[4~6]。
热激蛋白(Heat shock protein, HSP)是一类在生物体受到高温等逆境胁迫后大量表达的蛋白,用以协助蛋白质正确折叠,保护胞内蛋白质免受应激损伤,进而维持生物体的正常生理活动[7]。按分子量大小可将热激蛋白家族分为以下几族:HSP100、HSP90、HSP70、HSP60、HSP40、小分子sHSPs以及泛素蛋白[8]。其中,HSP70是分布最广、保守性最高的一支热激蛋白家族,其家族成员分布在细胞内的各个部位[9]。HSP70基因家族各成员由 3个部分组成:含有ATP酶结合域(ATPase domain)的N-端保守区,又称为环核苷酸结合域(Nucleotide binding domain, NBD),分子量约为44 kDa;可变C-端区域,分子量约为10 kDa;紧连ATPase结构域的一段多肽结合部位,该段区域氨基酸序列相对保守,分子量约为15 kDa[10,11]。由于HSP110与HSP70家族具有高度同源的基因序列和结构特征,所以尽管具有相对较大的分子量,也将其归为 HSP70超家族[12]。相比于动物HSP70的研究,植物HSP70家族才刚刚起步。已知在拟南芥(Arabidopsis thaliana)全基因组中共编码18个HSP70基因家族成员,其中14个只属于HSP70,另有4个为HSP110家族成员[13]。菠菜(Spinacia oleracea L.)中至少有12个HSP70基因[14],水稻(Oryza sativa)全基因组共含有32个HSP70家族成员[15]。
HSP70基因根据表达情况可以分为组成型和诱导型,组成型HSP70基因在植物体内普遍表达,可作为看家基因,而诱导型HSP70基因则只在受到外界刺激或特定的发育需要时大量表达[16]。植物HSP70的表达受高温等非生物胁迫的诱导[13,15],也受病原体入侵的调控,同时与植物发育相关[17]。朱一超等[18]应用cDNA芯片技术进行纤维伸长发育期的基因表达研究,发现在棉纤维发育过程中 HSP70基因在李氏超短纤维突变体材料中的表达量低于野生型材料。同时,在蛋白质水平上,也有研究发现HSP70互作蛋白在棉纤维发育过程中表达上调[19]。由此可知,热激蛋白70家族对棉纤维的发育起着重要作用。本研究利用生物信息学方法,对雷蒙德氏棉全基因组的HSP70基因进行了预测和系统进化分析,并对其在棉纤维发育过程中的基因表达进行分析,旨在为棉花优质、高产等重要农艺性状提供候选基因,为基因组水平上的棉花分子育种工作提供研究基础。
1 材料和方法
1.1 雷蒙德氏棉及其他物种 HSP70基因家族的数据获取
雷蒙德氏棉、可可(Theobroma cacao)、二穗短柄草(Brachypodium distachyon)、小立碗藓(Physcomitrella pattens)、卷柏(Selaginella moellendorffii)及火炬松(Pinus taeda)基因组及蛋白质序列均来自于Phytozome V9.0 (http://www.phytozome.net)数据库。根据文献下载拟南芥18个HSP70家族基因[13],利用隐马尔可夫模型(Hidden Markov Model, HMM)在雷蒙德氏棉全蛋白质序列数据库中搜索HSP70预测基因,所有预测基因序列在 SMART (http://smart. embl-heidelberg.de)和 InterProScan (http://www.ebi. ac.uk/interpro)、Pfam(http://pfam.sanger.ac.uk/)数据库中进行HSP70蛋白保守结构域的检查,E值设定为< e−20,其他参数为默认值。用同样方法获得可可、小立碗藓、卷柏和火炬松的HSP70家族序列。利用水稻HSP70家族相关文献上的序列获得单子叶植物二穗短柄草 HSP70基因家族成员[15],方法如上述。莱茵衣藻(Chlamydomonas reinhardtii)HSP70基因根据Renner等[20]报道的序列登录号下载。
1.2 基因复制共线性分析
利用BLAST软件将所获得的GrHSP70基因重新在基因组序列中定位,采用in-house python script软件对其分布情况进行直观展现,遵循其在染色体上的位置顺序将雷蒙德氏棉30个HSP70基因编号为 GrHSP70-1至 GrHSP70-24和 GrHSP110-1至GrHSP110-6。参照 Maher等[21]方法对 GrHSP70进行基因倍增模式分析。当几个HSP70基因位于同一或相邻的基因间区域(Intergenic region),则这些HSP70基因为串联重复(Tandem duplication)产物。取GrHSP70基因上、下游各30个蛋白质编码基因,将每一个蛋白质编码基因与雷蒙德氏棉基因组数据库中的蛋白质序列进行BLASTP比对,E值设定为<0.001,获取与该蛋白质最佳非自我匹配基因(The best no-self matches)。利用MCScanX软件对HSP70基因家族成员所在基因组区段进行基因复制共线性分析[22]。
1.3 HSP70蛋白序列的进化分析和基序分布
HSP70蛋白序列的多重比对利用ClustalX 1.81完成,参数为默认值。将生成的“.aln”文件导入MEGA 5.0,采用邻接法(Neighbor-joining)构建系统进化树,Bootstrap重复次数(Replications)设置为“500”[23]。
1.4 雷蒙德氏棉HSP70基因结构分析
利用GrHSP70基因的编码区序列(CDS)与全基因组序列比对,在 PIECE网站上(http://wheat.pw. usda.gov/piece/GSDraw.php)得到各基因的内含子、外显子的个数和排布情况。HSP70基因亚细胞定位预测采用WoLFPSORT(http://wolfpsort.seq.cbrc.jp/)、Softberry(http://www.softberry.com/)和 TargetP(http: //www.cbs.dtu.dk/services/TargetP/)数据库,综合上述3个软件的预测结果进行基因亚细胞定位分析。
1.5 棉纤维不同发育时期的表达分析
1.5.1 深度测序表达谱的获得与分析
陆地棉标准系 TM-1纤维和胚珠不同时期的深度测序表达谱下载于 NCBI网站 SRA数据库(http: //www.ncbi.nlm.nih.gov/sra/)。测序原始数据Fastq文件登录号分别为0 dpa(days post anthesis):SRX139593;3 dpa:SRX139594;7 dpa::SRX139595;15 dpa:SRX139596。利用In house perl script 分析HSP70基因家族每个基因的表达量。利用层次聚类算法分别分析HSP70基因在棉纤维和胚珠不同发育时期的表达数据。通过Cluster3.0展现层次聚类结果。
1.5.2 棉纤维不同发育时期的RNA提取及反转录
采用改良的CTAB法提取植物总RNA[24],样品分别为陆地棉TM-1开花当天0 dpa和3 dpa的胚珠、7 dpa和15 dpa纤维。粗提的RNA用DNase-Ⅰ(TaKaRa)进行处理,确保没有基因组DNA的污染。消化后的RNA样品采用Nanodrop ND-1000分光光度计进行定量,利用A260/A280nm和A260/A230nm的比值对样品质量进行鉴定。每个样品都采用 2 μg的RNA总量,运用反转录酶M-MLV(TaKaRa)完成cDNA的合成。采用HSP70基因特异性引物进行RT-PCR扩增,验证深度测序表达谱的分析结果。
1.5.3 RT-PCR结果检测
将上述方法得到的cDNA模板进行表达分析,分别选取HSP70-11、HSP70-12、HSP70-20、HSP70-24和HSP110-6基因,对其在棉纤维发育不同时期的表达进行实时定量PCR检测。PCR引物采用Primer 5.0软件设计,在基因序列 3′端特异性区域设计引物,扩增片段约为200 bp左右,由上海华大基因合成,所用引物序列见表 1。根据棉花看家基因泛素蛋白基因(Ubiquitin, UBQ)作为荧光定量 PCR扩增反应的内参,按照 2-ΔΔCt法计算基因的相对表达值[25]。实验中每种材料采集3组重复样品,进行3次重复提取RNA,每个样品的RNA需进行3次重复,共计9组实验结果,计算其平均值和标准差。

表1 PCR扩增引物
2 结果与分析
2.1 雷蒙德氏棉基因组中的HSP70基因分析
利用已知的拟南芥18个HSP70基因作为模板,运用HMMER软件在雷蒙德氏棉全基因组中共检索到30个GrHSP70基因,经过BLAST和结构域分析,确定这 30个基因均具有热激蛋白家族的保守蛋白结构域。其中,24个属于HSP70亚家族,按其在染色体上的位置分布,将这24个HSP70基因依次命名为:GrHSP70-1~GrHSP70-24,另外 6个基因归为HSP110/SSE亚家族,命名为GrHSP110-1~GrHSP110-6 (表2)。根据HSP70基因C-端的不同基序,进行亚细胞定位预测(表2)[14]。发现GrHSP70-1、GrHSP70-3、GrHSP70-4、GrHSP70-7、GrHSP70-9、GrHSP70-10、GrHSP70-13、GrHSP70-17、GrHSP70-18、GrHSP70-19、GrHSP70-20、GrHSP70-22和GrHSP70-24定位在细胞质中;GrHSP70-2、GrHSP70-5、GrHSP70-11、GrHSP70-12、GrHSP70-23定位于内质网中。GrHSP70-6、GrHSP70-8和GrHSP70-21定位于质体内。GrHSP70-14、GrHSP70-15、GrHSP70-16定位于线粒体中。将雷蒙德氏棉HSP70家族在拟南芥和水稻中的同源基因进行比较,可以发现它们具有相似的亚细胞定位情况[13,15]。
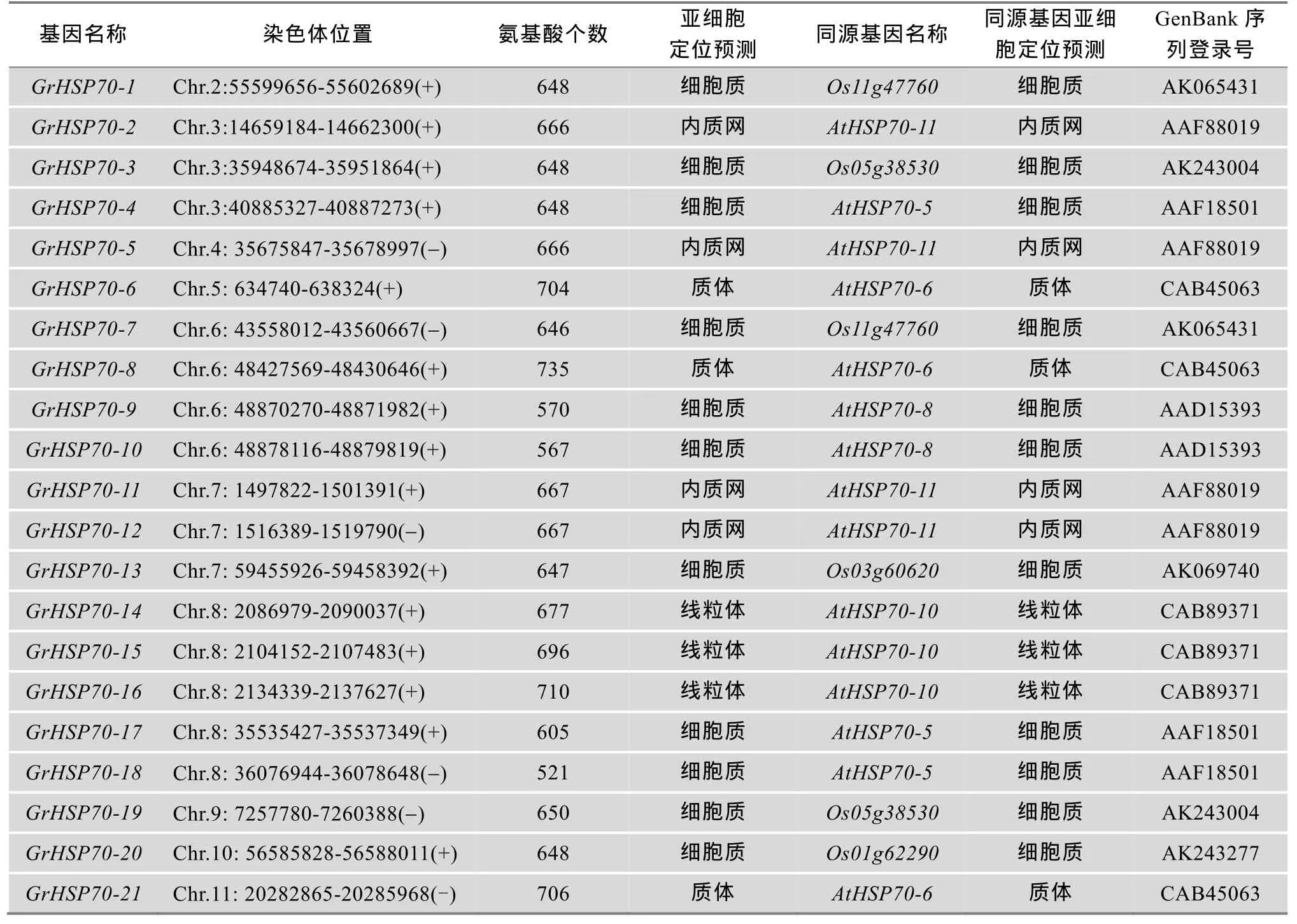
表2 雷蒙德氏棉HSP70基因家族基因组分析

续表2
2.2 GrHSP70基因家族序列特征和基序分布
将雷蒙德氏棉30个热激蛋白70家族的蛋白质序列进行系统进化分析,可以将其分为2个亚家族:HSP70亚家族和HSP110亚家族(图 1a)。每个亚家族内的基因按照不同的亚细胞定位结果,HSP70中的基因可分为4组(图1:A~D组),分别定位于细胞质、内质网、质体和线粒体;HSP110亚家族集中分布于细胞质和内质网中。分析这些基因氨基酸序列和其对应的DNA及mRNA序列,可得出基因结构图(图1b)。HSP110亚家族成员个数较少,具有基因序列长和内含子数目多的特点。HSP70亚家族成员个数较多,其中:A组的HSP70基因特点为均定位于细胞质中,基因中内含子数目少;B组为内质网类型,含有5个HSP70基因,核苷酸序列结构比A组复杂,平均具有8个外显子;C组和E组分别对应着质体类型和线粒体类型,这两个亚家族进化关系较近,同属于一个大的分支。

图1 GrHSP70家族系统进化树和基因结构分析a:雷蒙德氏棉HSP70家族系统进化分析;b:雷蒙德氏棉HSP70家族基因外显子-内含子的排布图。外显子和内含子分别用方框和黑线表示。
2.3 HSP70基因在二倍体棉花D组染色体上的分布
HSP70基因在棉花二倍体 D组的13条染色体上并不是均匀分布的,30个基因的具体定位情况见图2。其中,1号和12号染色体上未检测到HSP70基因,棉花第6号、第8号和第11号染色体上HSP70基因分布最多,均具有5个HSP70基因。HSP70基因在雷蒙德氏棉染色体上具有成簇分布的现象,在Phytozome数据库中通过 Gbrowse查看成簇分布的HSP70基因家族成员与侧翼蛋白质编码基因的位置关系。同一家族的不同成员如果位于同一个或相邻的基因间区域,则这些成员为串联重复关系[26]。按照此标准,雷蒙德氏棉HSP70基因家族有7个成员形成了3个串联重复组对(表3)。从Phytozome数据库分别获取雷蒙德氏棉HSP70基因家族各成员侧翼30个蛋白质编码基因,利用MCScanX软件对HSP70家族成员所在基因组区段进行基因复制共线性分析。由图3可知,部分雷蒙德氏棉HSP70基因家族成员之间存在显著的共线性关系,如 HSP70-2与HSP70-5、HSP110-3与HSP110-6,这些HSP70基因所定位的染色体区段是基因组进化过程中染色体大片段重复的产物,即位于染色体倍增块(Duplicated block)上。分析表明,共有15个HSP70基因家族成员位于 6对染色体倍增块上(表 3)。在HSP70基因的6对倍增块中还存在着交叉匹配的现象,即一个染色体片段同时与2个或3个染色体片段构成倍增块。例如:HSP70-1、HSP70-7、HSP70-13和HSP70-19倍增块,分别对应第2、6、7和9号染色体;HSP70-6、HSP70-8和HSP70-21倍增块,分别位于第5、6和11号染色体。这些倍增块的产生可能是由于染色体多次复制。
2.4 HSP70基因的系统进化分析
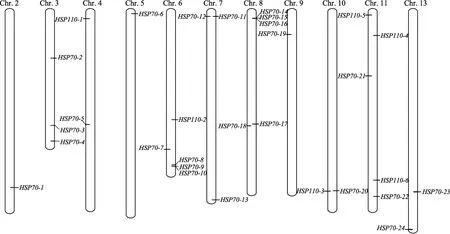
图2 HSP70基因在雷蒙德氏棉染色体上的分布情况
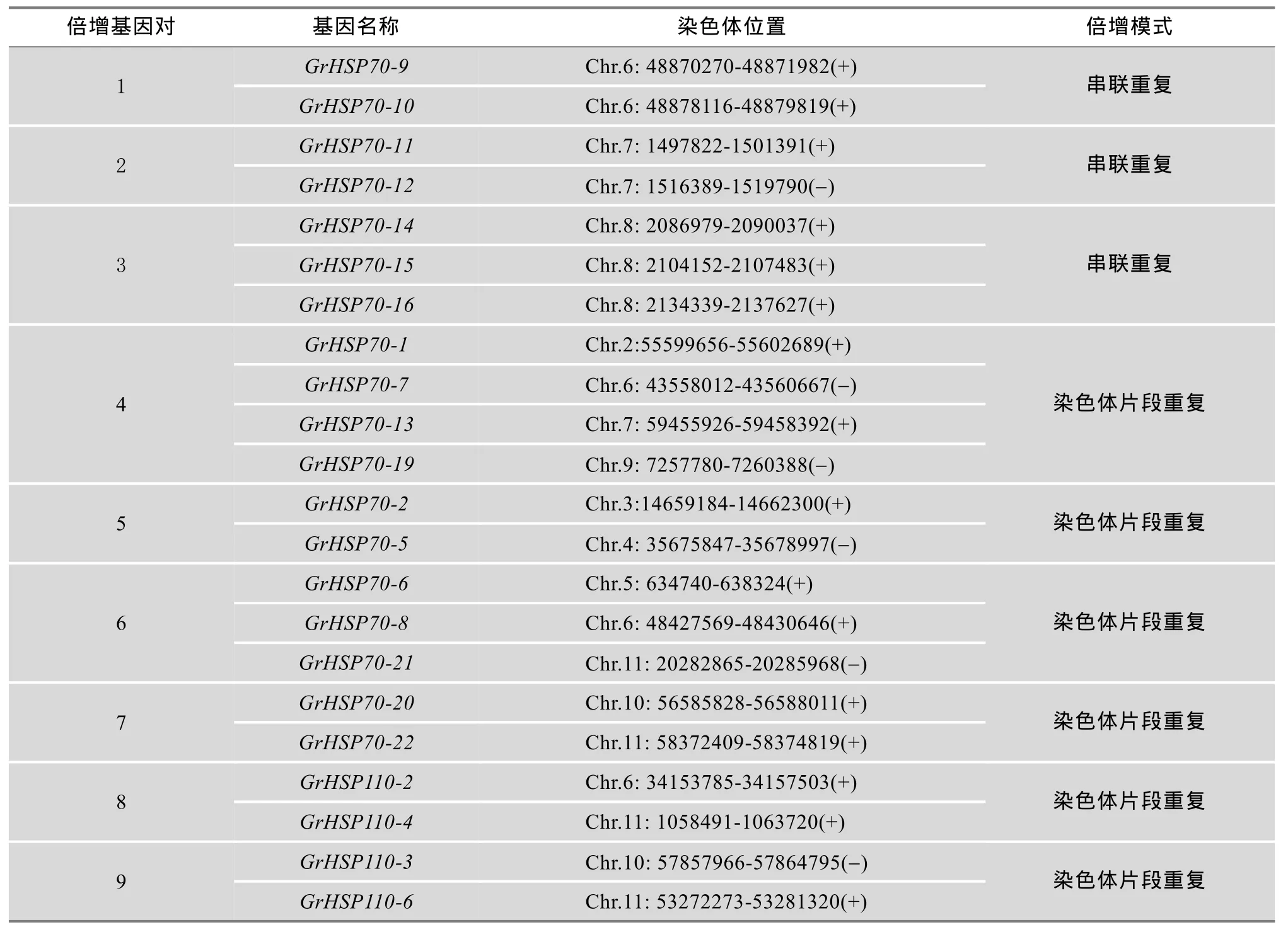
表3 GrHSP70基因倍增模式分析表
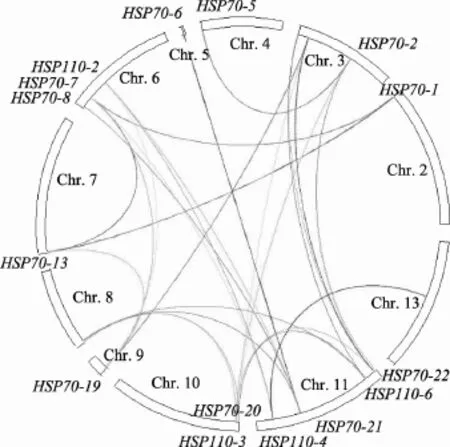
图3 雷蒙德氏棉HSP70基因在染色体上的共线性分析
为了进一步研究HSP70基因在不同物种中的进化情况,运用HMMER软件在可可、二穗短柄草、小立碗藓、卷柏和火炬松等物种的全基因组中搜索HSP70基因家族,加上文献中下载到的莱茵衣藻、拟南芥和水稻的HSP70基因,将这9个物种的蛋白质序列进行系统进化分析[13,15](图4)。根据基因进化关系将雷蒙德氏棉HSP70分为HSP70和HSP110两个亚家族,在每个亚家族中根据基因的亚细胞定位情况又可将各亚家族基因细分(表 4),分类结果与已报道的其他物种的HSP70相类似。其中莱茵衣藻的HSP70家族成员最少,分别具有5个HSP70亚族成员和2个HSP110亚族成员(系统进化树中用红色三角标注),二穗短柄草的 HSP70家族成员最多,有26个HSP70亚族和8个HSP110亚族。从表4可以发现,HSP70基因各亚组的分化发生在单细胞植物衣藻形成之前,从最简单的单细胞藻类到进化较高级的单子叶植物,都具有完整的HSP70家族成员种类。比较不同物种HSP70基因家族的亚组成员数可以发现,在低等植物衣藻中,各亚组中基本都只具有1到2个HSP70基因成员,而在较高等的植物中,HSP70基因个数有所增加,但不同亚组成员数增加的比例不同,总体来说,胞质型HSP70数目比其他亚组成员数增加较多,典型代表为火炬松,具有25个细胞质型HSP70基因,而细胞器特异性的HSP70基因个数几乎与莱茵衣藻相同。由系统进化树可知,线粒体型HSP70的进化关系基本符合植物的进化关系,由莱茵衣藻、小立碗藓、卷柏为代表的低等植物形成一个外群体(Out group),裸子植物火炬松与被子植物形成两个分支,同时,单双子叶植物分别聚类。相比线粒体型HSP70基因,其他亚组的HSP70的进化关系并不能明显的呈现生物进化关系,尤其是胞质型 HSP70。因此推测在基因复杂的复制过程中可能发生了基因丢失(Gene lost)或基因转换事件(Gene conversion)。
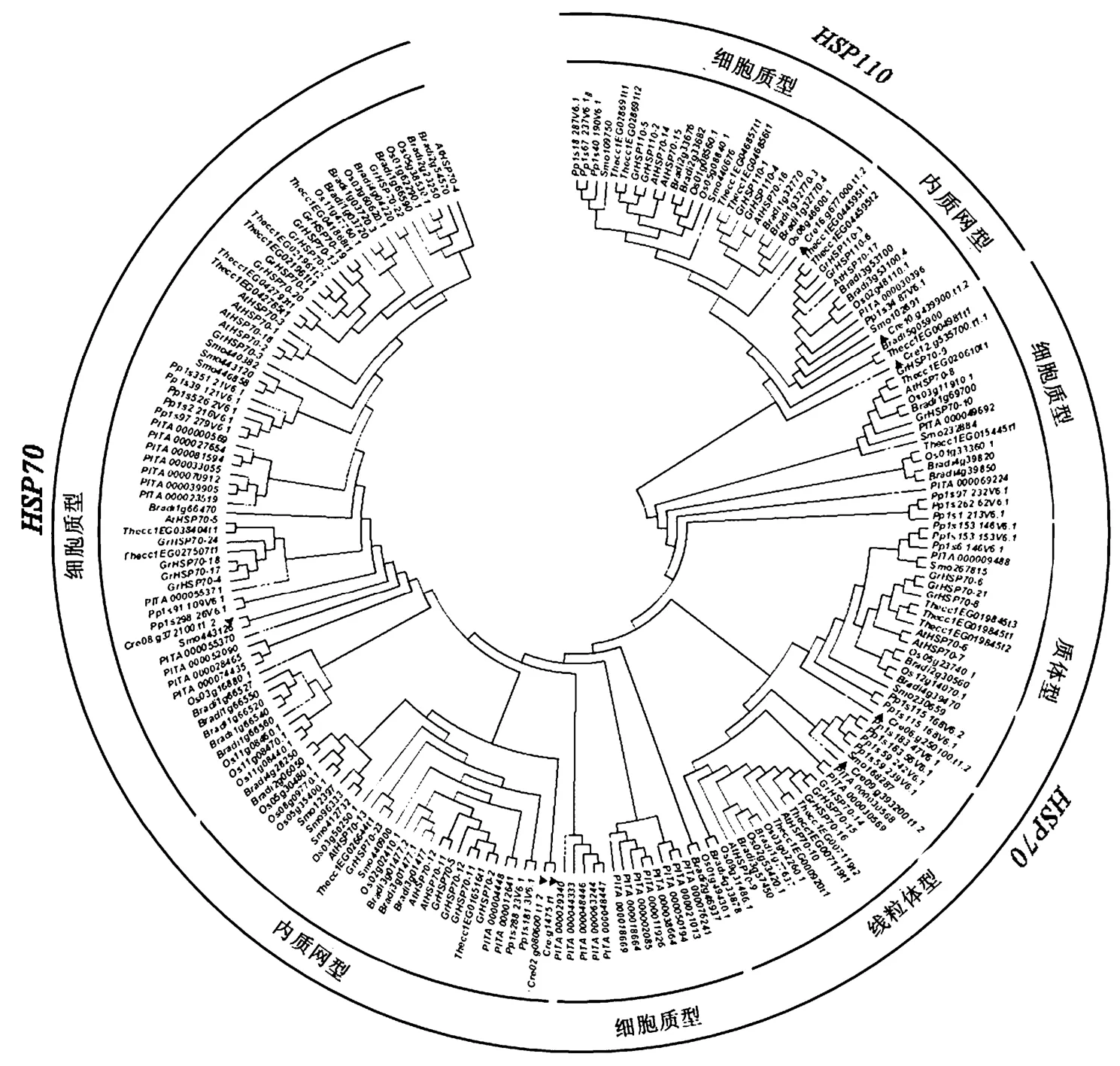
图4 9个物种的HSP70基因家族的系统进化分析9个物种分别为雷蒙德氏棉(Gr)、拟南芥(At)、水稻(Os)、可可(Thecc)、二柄短尾草(Bradi)、莱茵衣藻(Cre)、小立碗藓(Pp)、卷柏(Smo)和火炬松(PITA)。
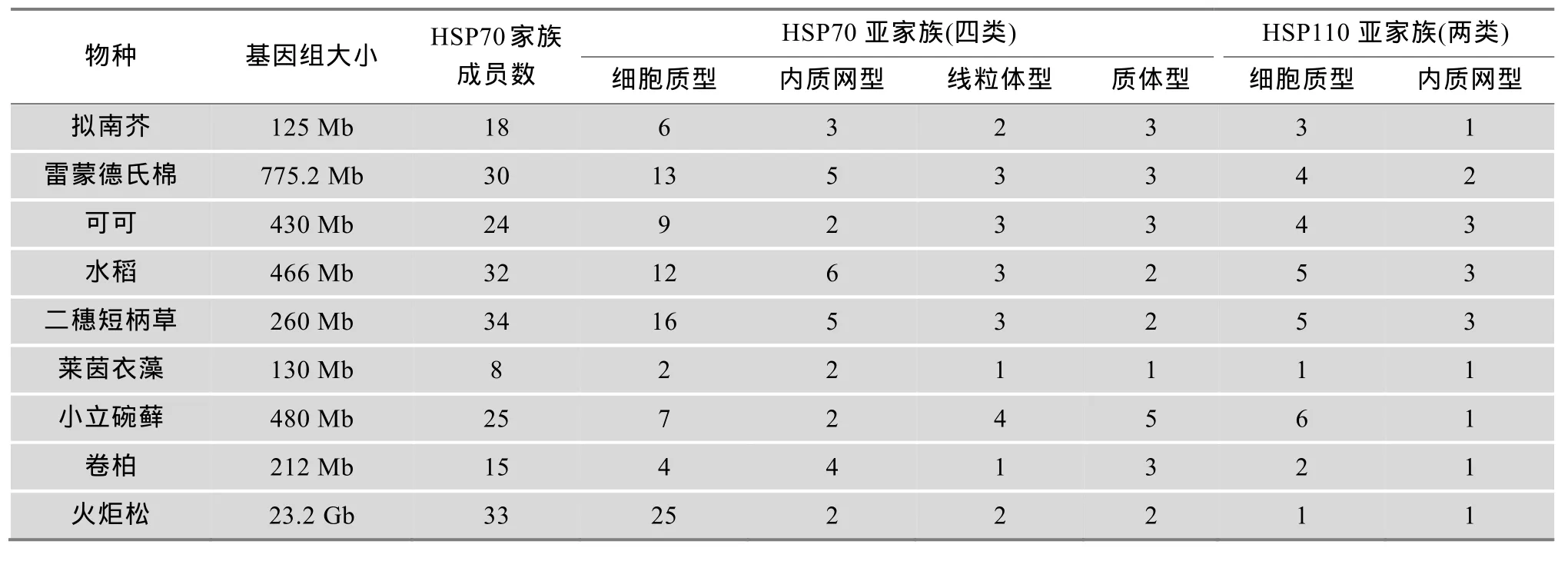
表4 各物种中HSP70基因家族成员统计
2.5 HSP70基因在陆地棉TM-1纤维发育不同时期的表达分析
由于四倍体陆地棉是世界上分布最广泛的棉种,研究HSP70基因在陆地棉纤维发育过程中的表达情况更具有实际意义。对陆地棉标准系 TM-1中GrHSP70家族的同源基因进行表达分析,深度测序数据选择棉纤维发育的4个时期(0 dpa、3 dpa、7 dpa及15 dpa)。由图5 A可知,HSP70-4、10、17、18、22和 23未在胚珠或纤维中检测到表达。同时选取雷蒙德氏棉和陆地棉不同组织部位和时期的深度测序数据进行表达分析,除HSP70-22、23在雷蒙德氏棉叶片中表达外,其他 4个基因仍未检测到表达,推测这 4个 HSP70基因为假基因。HSP70-2、HSP70-11、HSP70-13、HSP70-20及HSP110-3在这4个纤维发育的不同时期都高表达,推测这几个基因在棉纤维发育各个过程中均起重要作用。同时,和对照0 dpa的胚珠表达相比(图5 B),在0 dpa表达量高,而之后逐渐下调的基因有 HSP70-2、11、12及HSP110-4、6;在0 dpa表达量低,在之后的发育时期表达量上升的基因有HSP70-24、20、13、8、19。其他 HSP70基因的表达在发育的某一时期具有特异性,如 HSP70-8在 7 dpa表达量较高,HSP70-3在3 dpa高表达。
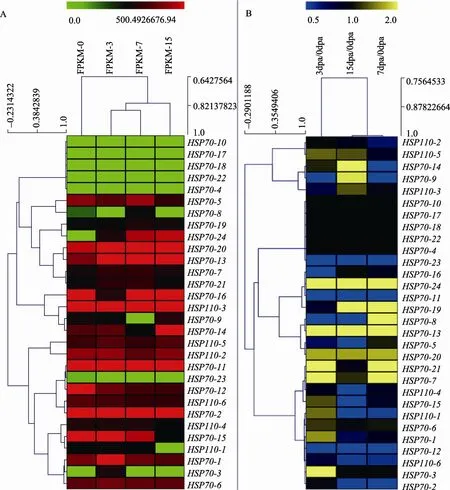
图5 GrHSP70基因家族在陆地棉中同源基因纤维不同发育时期的表达分析A:利用分层聚类算法分析HSP70基因在4个棉纤维发育阶段(0 dpa、3 dpa、7 dpa、15 dpa)的深度测序数据,通过树形视图呈现分层聚类结果。红色表示表达量高,绿色表示表达量较低。B:将各HSP70基因0 dpa的表达数据作为对照,其他3个时期(3 dpa、7 dpa、15 dpa)的芯片数据与其比较得到分层聚类结果。黄色表示表达量相对于0 dpa上调,黑色表示表达量不变,蓝色表示表达下调。
选取表达量变化趋势较明显的几组HSP70基因进行RT-PCR验证,根据2-ΔΔCt法计算基因的相对表达值,结果如图6所示,与0 dpa相比,HSP70-11、HSP70-12与HSP110-6在3 dpa、7 dpa和15 dpa表达呈下调趋势,而HSP70-20和HSP70-24表达量与0 dpa相比逐渐升高。这些基因的表达趋势与深度测序分析所得结果相同,说明深度测序的结果真实可靠。
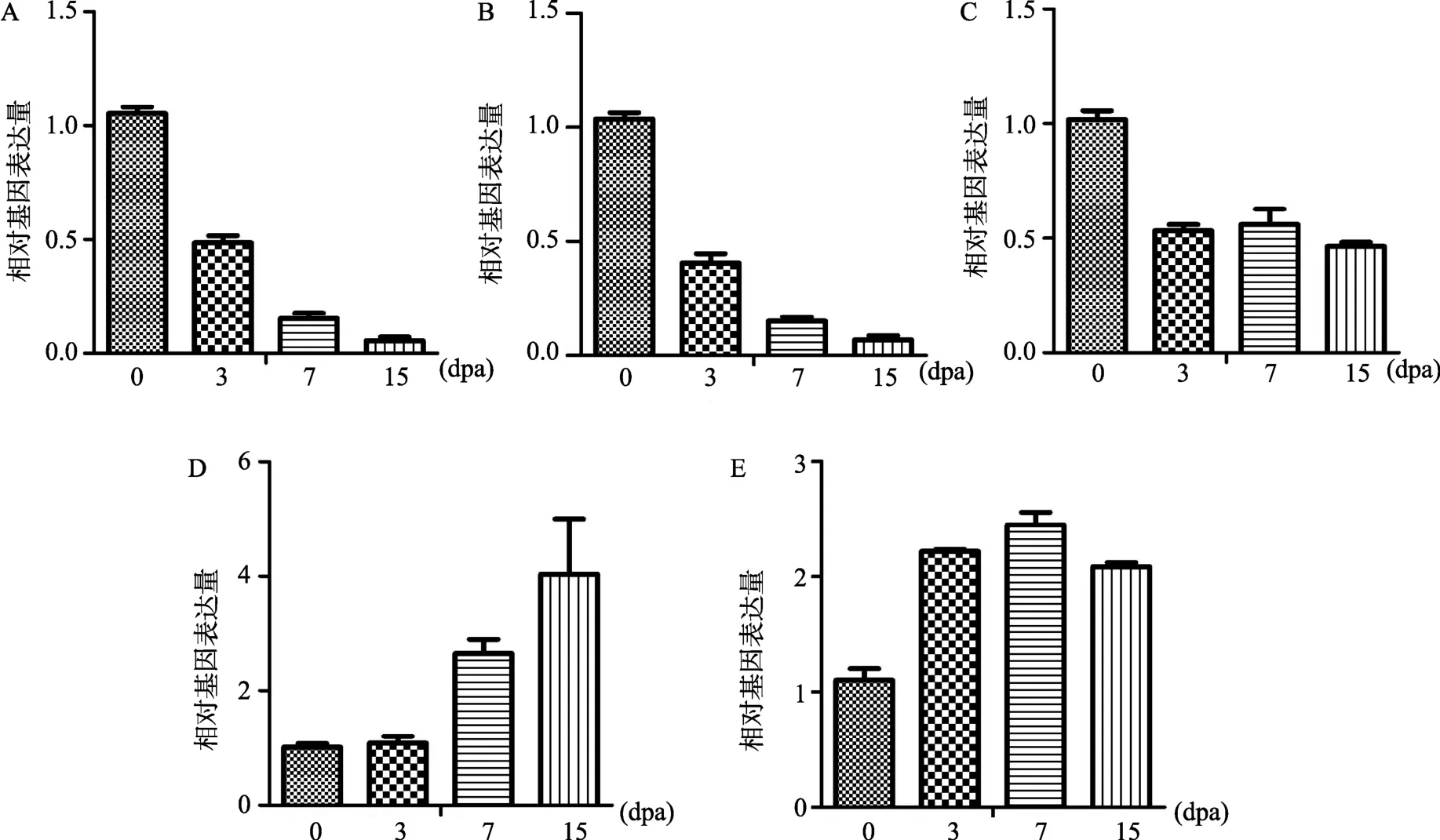
图6 HSP70基因RT-PCR表达分析A:HSP70-11;B:HSP70-12;C:HSP110-6;D:HSP70-24;E:HSP70-20。
3 讨 论
本文在基因组水平上对雷蒙德氏棉HSP70基因进行了鉴定和功能分析。雷蒙德氏棉HSP70超家族共有30个成员,其中包括24个HSP70基因和6个HSP110基因,约为拟南芥中该基因家族成员个数的2倍,这说明雷蒙德氏棉染色体相比于拟南芥染色体,在进化过程中经历了多次全基因组复制事件[13]。根据C-端的基序类型以及WOLF网站预测所得到的24个HSP70基因的亚细胞定位,将棉花HSP70基因进行分组,由此所得的4个HSP70亚家族与根据系统进化树的分支进行的分组情况相符,表明该家族基因的亚细胞定位分化具有高度保守性。HSP110蛋白与HSP70蛋白的区别主要在于具有 N-端ATP酶结合域和 C-端之间的插入区段或 C-端的延长区域,较大的分子质量使HSP110自成一个亚家族[12]。比较GrHSP70基因的内含子、外显子排布情况及基序的分布情况,可以发现,同一亚组中的各基因具有类似的基因结构,这些相似度极高的序列与基因组进化过程中的串联重复、随机重复与插入等现象有关,发生在各亚家族分化之后,在基因家族的进化和扩充过程中起到重要作用[27]。雷蒙德氏棉为现有四倍体栽培棉品种的 D组染色体祖先,研究HSP70基因的多次复制现象可以帮助人们更好地了解棉花基因组多倍化的形成过程。
雷蒙德氏棉的基因组经历了全基因组重复、染色体重排和串联重复等复杂的进化过程[5]。本研究结果表明,染色体大片段复制和串联重复在 HSP70基因家族的扩增中起到重要作用。在雷蒙德氏棉中共有22个HSP70基因形成9个基因对,其中线粒体型 HSP70基因的扩增方式为串联重复,质体型HSP70亚组的扩增为染色体复制形成的,胞质型和内质网型HSP70基因的扩增为串联重复和染色体片段复制的共同产物。从棉纤维发育不同时期的表达数据来看,这些由同一基因祖先扩增而来的基因对,并不具有相似的表达模式,说明在进化过程中其表达特性发生趋异分化现象。
雷蒙德氏棉全基因组测序的结果表明,双子叶植物可可与棉花的亲缘关系较近[4]。单个基因家族HSP70的研究结果同样表明,可可和棉花具有较近的亲缘关系。将棉花、水稻、拟南芥、可可、二柄短尾草、莱茵衣藻等9个HSP70基因家族进行系统发生分析,发现HSP70不同亚组的分化发生在单细胞藻类植物形成之前。本文所列举的 9种代表植物中均具有HSP70家族全部的亚组种类,这与所报道的HSP70基因家族从原核生物到真核生物都高度保守的特征相吻合[27,28]。所列各物种的HSP70基因家族成员个数并不与基因组大小完全成正比,说明HSP70基因在各物种中的进化和复制具有多样性。众所周知,多倍体化在陆生植物的进化过程中多次发生,并且这些多倍体事件对植物基因家族的扩充起到了重要作用。HSP70基因家族从单细胞莱茵衣藻的8个成员扩充到二穗短柄草的33个成员有可能是多倍体事件的产物。Blanc和Wolfe在拟南芥中发现胞质类蛋白质比细胞器特异性蛋白质更容易保留复制形成的新成员[29]。这一结论也适用于本文列举的9种植物的HSP70基因家族进化情况。在陆生植物的漫长进化过程中,细胞器特异性的HSP70保持着相对稳定的数量,而胞质型HSP70则在各物种进化的选择压力下发生了不同程度的多倍化[20]。胞质HSP70是重要的热激诱导表达基因,对生物体的耐热性具有重要作用。关于果蝇(Drosophila melanogaster)多拷贝胞质HSP70对其适应高温胁迫的作用已进行了深入的研究[30,31]。相比于莱茵衣藻(2个胞质HSP70),推测裸子和被子植物中数量较多的胞质HSP70可以提高陆生植物的耐热性。
近年来,对植物HSP70功能的研究增多。目前已从拟南芥、烟草(Nicotiana tabacum L.)、玉米(Zea mays)等植物中获得了多个该家族成员,发现HSP70基因在植物细胞中主要起到分子伴侣功能,除参与新生肽链的折叠、损伤蛋白的降解外,还负责一些前体蛋白向叶绿体[32]、内质网和线粒体[33]的转运。May等[34]及惠颖等[35]在研究可能在棉花纤维起始和伸长发育阶段起调控作用的14-3-3蛋白质时,发现叶绿体HSP70蛋白(gi|297734242)与14-3-3蛋白互作,二者结合形成复合体共同协调叶绿体或线粒体蛋白质的跨膜运输,该HSP70蛋白与本文中GrHSP70-21同源性最高。Pang等[36]在对10 dpa的野生型陆地棉和徐州 142无纤维突变体的胚珠进行比较蛋白质组学分析,分离到多个属于 HSP70家族的蛋白(FJ415194;FJ415195;FJ415196;FJ415199)在野生型中表达量显著高于无纤维突变体,这些基因序列分别与 GrHSP70-13、GrHSP70-1、GrHSP70-20、GrHSP70-22具有较高同源性。由此,推测一些HSP70基因家族成员在棉纤维的发育过程中对蛋白质合成和运输起到调控作用,当这些基因表达表达下调时,会影响棉纤维的正常发育,导致短绒或无纤维。
本文对雷蒙德氏棉HSP70家族在陆地棉TM-1中的同源基因进行表达分析,发现大量HSP70基因广泛参与到棉纤维发育这一复杂的生理过程中。在纤维发育的不同时期,特异性表达的HSP70基因各不相同,在起始阶段表达量高的HSP70基因可能涉及到纤维原始细胞的分化和突起过程,在中期表达量显著的基因与其他基因协同作用,促进纤维初生壁伸长和次生壁增厚,而在后期表达量上升的HSP70基因则协助细胞脱水成熟。本文利用深度测序数据对HSP70家族基因在棉纤维发育过程中的作用进行了初步探索,而其如何参与每一发育过程的分子调控机制还需要进一步的研究。
[1] Xu ZY, Kohel RJ, Song GL, Cho J, Alabady M, Yu J, Koo P, Chu J, Yu SX, Wilkins TA, Zhu YX,Yu JZ. Gene-rich islands for fiber development in the cotton genome. Genomics, 2008, 92(3): 173-183.
[2] Ferguson DL, Turley RB, Triplett BA, Meredith WR. Comparison of protein profiles during cotton (Gossypium hirsutum L.) fiber cell development with partial sequences of two proteins. J Agric Food Chem, 1996, 44(12): 4022-4027.
[3] 张辉, 汤文开, 谭新, 龚路路, 李学宝. 棉纤维发育及其相关基因表达调控研究进展. 植物学通报, 2007, 24(2): 127-133.
[4] Wang KB, Wang ZW, Li FG, Ye WW, Wang JY, Song GL, Yue Z, Cong L, Shang HH, Zhu SL, Li Q, Yuan YL, Lu CR, Wei HL, Gou CY, Zheng ZQ, Yin Y, Zhang XY, Liu K, Wang B, Song C, Shi N, Kohe RJ, Percy RG, Yu JZ, Zhu YX, Wang J, Yu SX. The draft genome of a diploid cottonGossypium raimondii. Nature Genetics, 2012, 44(10): 1098-1103.
[5] Lin LF, Pierce GJ, Bowers JE, Estill JC, Compton RO, Rainville LK, Kim C, Lemke C, Rong JK, Tang HB, Wang XY, Braidotti M, Chen AH, Chicola K, Collura K, Epps E, Golser W, Grover C, Ingles J, Karunakaran S, Kudrna D, Olive J, Tabassum N, Um E, Wissotski M, Yu Y, Zuccolo A, ur Rahman M, Peterson DG, Wing RA, Wendel JF, Paterson AH. A draft physical map of a D-genome cotton species (Gossypium raimondii). BMC Genomics, 2010, 11(1): 395.
[6] Paterson AH, Wendel JF, Gundlach H, Guo H, Jenkins J, Jin D, Llewellyn D, Showmaker KC, Shu S, Udall J, Yoo MJ, Byers R, Chen W, Doron-Faigenboim A, Duke MV, Gong L, Grimwood J, Grover C, Grupp K, Hu G, Lee TH, Li J, Lin L, Liu T, Marler BS, Page JT, Roberts AW, Romanel E, Sanders WS, Szadkowski E, Tan X, Tang H, Xu C, Wang J, Wang Z, Zhang D, Zhang L, Ashrafi H, Bedon F, Bowers JE, Brubaker CL, Chee PW, Das S, Gingle AR, Haigler CH, Harker D, Hoffmann LV, Hovav R, Jones DC, Lemke C, Mansoor S, ur Rahman M, Rainville LN, Rambani A, Reddy UK, Rong JK, Saranga Y, Scheffler BE, Scheffler JA, Stelly DM, Triplett BA, Van Deynze A, Vaslin MF, Waghmare VN, Walford SA, Wright RJ, Zaki EA, Zhang T, Dennis ES, Mayer KF, Peterson DG, Rokhsar DS, Wang X, Schmutz J. Repeated polyploidization of Gossypium genomes and the evolution of spinnable cotton fibres. Nature, 2012, 492(7429): 423-427.
[7] Large AT, Goldberg MD, Lund PA. Chaperones and protein folding in the archaea. Biochem Soc Trans, 2009, 37(Pt 1): 46-51.
[8] Parsell DA, Lindquist S. The function of heat shock proteins in stress tolerance: degradation and reactivation of damaged proteins. Annu Rev Biochem, 1993, 27(1): 437-496.
[9] Kiang JG, Tsokos GC. Heat shock protein 70 kDa: molecular biology, biochemistry, and physiology. Pharmacol Ther, 1998, 80(2): 183-201.
[10] Bukau B, Weissman J, Horwich A. Molecular chaperones and protein quality control. Cell, 2006, 125(3): 443-451.
[11] Dragovic Z, Broadley SA, Shomura Y, Bracher A, Hartl FU. Molecular chaperones of the Hsp110 family act as nucleotide exchange factors of Hsp70s. EMBO J, 2006, 25(11): 2519-2528.
[12] Liu QL, Hendrickson WA. Insights into Hsp70 chaperone activity from a crystal structure of the yeast Hsp110 Sse1. Cell, 2007, 131(1): 106-120.
[13] Lin BL, Wang JS, Liu HC, Chen RW, Meyer Y, Barakat A, Delseny M. Genomic analysis of the Hsp70 superfamily in Arabidopsis thaliana. Cell Stress Chaperones, 2001, 6(3): 201-208.
[14] Guy CL, Li QB. The organization and evolution of the spinach stress 70 molecular chaperone gene family. Plant Cell, 1998, 10(4): 539-556.
[15] Sarkar NK, Kundnani P, Grover A. Functional analysis of Hsp70 superfamily proteins of rice (Oryza sativa). Cell Stress Chaperones, 2013, 18(4): 427-437.
[16] Sung DY, Kaplan F, Guy CL. Plant Hsp70 molecular chaperones: protein structure, gene family, expression and function. Physiol Plant, 2002, 113(4): 443-451.
[17] Noël LD, Cagna G, Stuttmann J, Wirthmüller L, Betsuyaku S, Witte CP, Bhat R, Pochon N, Colby T, Parker JE. Interaction between SGT1 and cytosolic/nuclear HSC70 chaperones regulates Arabidopsis immune responses. Plant Cell, 2007, 19(12): 4061-4076.
[18] 朱一超, 张天真, 贺亚军, 郭旺珍. 棉花纤维伸长发育期的基因表达分析. 作物学报, 2006, 32(11): 1656-1662.
[19] Zhao PM, Wang LL, Han LB, Wang J, Yao Y, Wang HY, Du XM, Luo YM, Xia GX. Proteomic identification of differentially expressed proteins in the Ligon lintless mutant of upland cotton (Gossypium hirsutum L.). J Proteome Res, 2009, 9(2): 1076-1087.
[20] Renner T, Waters ER. Comparative genomic analysis of the Hsp70s from five diverse photosynthetic eukaryotes. Cell Stress Chaperones, 2007, 12(2): 172.
[21] Maher C, Stein L, Ware D. Evolution of Arabidopsis microRNA families through duplication events. Genome Res, 2006, 16(4): 510-519.
[22] Wang YP, Tang HB, Debarry JD, Tan X, Li JP, Wang XY, Lee TH, Jin HZ, Marler B, Guo H, Kissinger JC, Paterson AH. MCScanX: a toolkit for detection and evolutionary analysis of gene synteny and collinearity. Nucleic Acids Res, 2012, 40(7): e49.
[23] Tamura K, Dudley J, Nei M, Kumar S. MEGA4: molecular evolutionary genetics analysis (MEGA) software version 4. 0. Mol Biol Evol, 2007, 24(8): 1596-1599.
[24] 胡根海, 喻树迅. 利用改良的 CTAB 法提取棉花叶片总RNA. 棉花学报, 2007, 19(1): 69-70.
[25] Livak KJ, Schmittgen TD. Analysis of relative gene expression data using real-time quantitative PCR and the 2-ΔΔCTmethod. Methods, 2001, 25(4): 402-408.
[26] Cannon SB, Mitra A, Baumgarten A, Young ND, May G. The roles of segmental and tandem gene duplication in the evolution of large gene families in Arabidopsis thaliana. BMC Plant Biol, 2004, 4: 10.
[27] Hu WH, Hu GC, Han B. Genome-wide survey and expression profiling of heat shock proteins and heat shockfactors revealed overlapped and stress specific response under abiotic stresses in rice. Plant Science, 2009, 176(4): 583-590.
[28] Bukau B, Horwich AL, Cycles H. The Hsp70 and Hsp60 review chaperone machines. Cell, 1998, 92(3): 351-366.
[29] Blanc G, Wolfe KH. Functional divergence of duplicated genes formed by polyploidy during Arabidopsis evolution. Plant Cell, 2004, 16(7): 1679-1691.
[30] Lerman DN, Feder ME. Naturally occurring transposable elements disrupt hsp70 promoter function in Drosophila melanogaster. Mol Biol Evol, 2005, 22(3): 776-783.
[31] Garbuz D, Evgenev MB, Feder ME, Zatsepina OG. Evolution of thermotolerance and the heat-shock response: evidence from inter/intraspecific comparison and interspecific hybridization in the virilis species group of Drosophila. I. Thermal phenotype. J Exp Biol, 2003, 206(14): 2399-2408.
[32] Waegemann K, Paulsen H, Soll J. Translocation of proteins into isolated chloroplasts requires cytosolic factors to obtain import competence. FEBS Letters, 1990, 261(1): 89-92.
[33] Deshaies RJ, Koch BD, Werner-Washburne M, Craig EA, Schekman R. A subfamily of stress proteins facilitates translocation of secretory and mitochondrial precursor polypeptides. Nature, 1988, 332(6167): 800-805.
[34] May T, Soll J. 14-3-3 proteins form a guidance complex with chloroplast precursor proteins in plants. Plant Cell, 2000, 12(1): 53-63.
[35] 惠颖, 王晋, 孙敬, 刘康, 唐灿明. 棉花Gh14-3-3L2 基因的分子克隆及其互作蛋白质的初步鉴定. 棉花学报, 2012, 24(4): 285-292.
[36] Pang CY, Wang H, Pang Y, Xu C, Jiao Y, Qin YM, Western TL, Yu SX, Zhu YX. Comparative proteomics indicates that biosynthesis of pectic precursors is important for cotton fiber and Arabidopsis root hair elongation. Mol Cell Proteomics, 2010, 9(9): 2019-2033.
(责任编委: 刘 宝)
Genome-wide analysis of HSP70 superfamily in Gossypium raimondii and the expression of orthologs in Gossypium hirsutum
Yuting Zhang1,2, Minhua Wang1,2, Jiadong Chen2, Junkang Rong2,3, Mingquan Ding2,3
1. The Nurturing Station for the State Key Laboratory of Subtropical Silviculture, Zhejiang Agricultural and Forestry University, Lin’an 311300, China;
2. The Key Laboratory for Quality Improvement of Agricultural Products of Zhejiang Province, School of Agriculture and Food Science, Zhejiang Agricultural and Forestry University, Lin’an 311300, China;
3.Institute of Cotton Research, Chinese Academy of Agricultural Sciences, State Key Laboratory of Cotton Biology, Anyang 455000, China
Heat shock 70 (HSP70) proteins are highly conserved molecular chaperones widely existed in the plant kingdomwhich are involved in cellular protein folding process. In this study, comprehensive evolutionary analyses of the Gossypium raimondii HSP70 gene family members are conducted and 30 HSP70 genes are identified. The gene structure, chromosome distribution, gene duplication and phylogenic evolution of this family are further analyzed. The results reveal that HSP70 family genes can be clustered into several major subgroups based on their sub-cellular locations, and the gene structures are relatively conserved in each subgroup. Both tandem duplications and chromosome segmental duplications are found to contribute to the expansion of HSP70 gene family. Evolutionary analysis of HSP70s in diverse species reveals that the differentiation of HSP70 subgroups occurred before the multi-cell and single-cell differentiation, with the cytoplasmic HSP70s multiple amplified. The expression pattern of HSP70 genes under series of fiber development stages indicates that many HSP70 genes may participate in fiber development processes including fiber initiation and elongation. This study provides the complete profiles of cotton HSP70 family genes for future study on their functions related to the molecular mechanisms of fiber development.
cotton fiber; HSP70 family; deep sequencing; gene structure; phylogenic analysis
2014-03-11;
2014-05-20
国家高技术研究发展计划项目(863计划)(编号:2011AA100202),国家自然科学基金项目(编号: 31200909),浙江省自然科学基金项目(编号: LQ12C06002)和棉花生物学国家重点实验室开放课题(编号:CB2013A02,CB2014B04)资助
张毓婷,在读硕士研究生,专业方向:棉花遗传与分子育种。E-mail:zhangyutingly@126.com
丁明全,讲师,研究方向:棉花遗传与分子育种。E-mail:mqding@bjfu.edu.cn
戎均康,教授,研究方向:棉花遗传与分子育种。E-mail:jkrong@yahoo.com
10.3724/SP.J.1005.2014.0921
时间: 2014-7-31 15:10:03
URL: http://www.cnki.net/kcms/detail/11.1913.R.20140731.1510.001.html

