异染色质相关蛋白TRIM28调控锌指蛋白和原钙黏蛋白家族基因的转录
张翔宇 卜佳晨
(中国科学院生物物理研究所,生物大分子国家重点实验室,北京 100101)
TRIM28 (TRIpartate motif‑containing protein 28),又称为KAP1(KRAB‑associated protein1)或者TIF1β(transcription intermediary factor 1β),它由N 端的RBCC 区域和C 端的PHD、BROMO 和NHD等结构域组成[1]。既往研究发现,TRIM28具有E3 活性,通过SUMO 通路招募SETDB1 和HP1等异染色质相关因子,在靶基因上建立H3K9me3修饰,抑制基因的转录[2]。此外,还有研究发现,TRIM28 通过调控RNA 聚合酶II 在Hspa1b 等 基 因启动子区域的转录延伸活性调控基因的表达[3]。
为了更深入地研究TRIM28的功能,本文通过建立Trim28 基因敲除细胞系,发现Trim28 主要抑制内源表达量较低基因的转录,进一步分析发现Trim28抑制锌指蛋白家族基因和原钙黏蛋白β家族基因的转录,ChIP‑seq 数据证明TRIM28 通过影响染色质高级结构的改变调控锌指蛋白家族基因和原钙黏蛋白β家族基因的转录。
原钙黏蛋白家族基因(protocadherin,PCDH)分为PCDHα、PCDHβ和PCDHγ 3个类别,在神经细胞中具有较高的表达水平。原钙黏蛋白对神经元突触的发育具有重要作用,能够影响神经细胞对“自我”和“非我”身份识别过程[4]。已有文章报道,染色质高级结构的改变能够影响原钙黏蛋白家族基因的表达[5‑6],但是对于特定的激活PCDHα基因、PCDHβ基因或者PCDHγ基因的转录活性机制并不清楚。
本文通过生物组学的方法首次发现了TRIM28与原钙黏蛋白家族基因转录调控之间的关系,并证明了两者之间的联系,对于TRIM28的分子机理的进一步的研究提供了新的思路。
1 材料与方法
1.1 Trim28基因敲除细胞系和Trim28回补细胞系的构建
根据实验室之前设计向导RNA(gRNA)的方法[7‑9],设计靶向Trim28基因第一个外显子起始密码子下游的两对小向导RNA(sgRNA),gRNAF1:5'‑caccgggcctccgcggcggcagcct‑3'和gRNAR1:5'‑aaa‑caggctgccgccgcggaggccc‑3';gRNAF2:5'‑caccggcg‑acccgagagggagcccc‑3'和gRNAR2:5'‑aaacggggctccc‑tctcgggtcgcc‑3'。 将 经 过 退 火 的 引 物 连 接 到pLentiCas9V2质粒上,原核培养提取质粒。根据转染试剂Vigofect 说明书转染质粒至HEK293F 细胞中,96 孔板分单克隆。使用DNA 提取液快速提取细胞DNA,DNA 提取液成分是:100 mmol/L Tris‑HCl、500 mmol/L KCl、0.01% 明 胶、0.45%NP‑40、0.45%吐温。设计引物检测阳性克隆,引物序列是Test F:5'‑agttggccgtgccgtagcagcgtcc‑3'和Test R:5'‑gccctaagcaggcactacaggccg‑3'。PCR 产 物送测序公司测序,测序结果和野生型相比较,有DNA序列的突变即是阳性克隆。
利用pCMV‑cTrim28‑mCherry 载体和转座子转染体系,在Trim28 基因敲除细胞系中共转Piggyback 转座酶表达载体,转座酶可促使pCMV‑cTrim28-mCherry 上相应Trim28cDNA 片段整合到转染的细胞基因组中,转座成功的细胞,通过CMV 启动子表达mCherry 荧光,使用流式细胞分选仪分选出对应的细胞。
1.2 mRNA反转录与实时荧光定量PCR
根据说明书推荐的使用方法,用TRIzol®裂解液(ThermoFisher,15596‑026)裂解细胞提取RNA,NanoDrop 2000C 分光光度计(Thermo Scientific 公司)测定RNA 浓度,采用HiScript®II RT SuperMix for qPCR试剂盒(南京诺唯赞生物公司,R122‑01)的操作步骤,大约1~2 μg RNA 作为模板,反转录完成后稀释10 倍。PCR 反应体系是:10 μl 2×Taq Pro Universal SYBR qPCR Master Mix(南京诺唯赞生物公司,Q712‑02),0.4 μl 10 mmol/L引物,5 μl cDNA模板,4.2 μl dd H2O。在7500 Fast Real‑Time PCR System(ABI 公司)仪器上按照预变性95℃30 s,循环反应95℃10 s,60℃30 s,40周期,溶解曲线95℃15 s,60℃60 s,95℃15 s 设定程序,进行荧光定量PCR反应。引物序列见表1。

Table 1 Primers for real-time quantitative PCR
1.3 免疫印迹
根据细胞的数量加入适量的细胞裂解液和蛋白酶抑制剂混匀后充分裂解细胞,细胞裂解液成分是:100 mmol/L Tris‑HCl pH 6.8,4% SDS,20%甘油,200 mmol/L DTT,0.2%溴酚蓝。利用蛋白质浓度检测试剂盒(碧云天公司,P0012S)测出各个样品浓度,将样品调至相同浓度,20 mA电泳1.5~2 h,250 mA 转PVDF 膜,一 抗 过 夜 孵 育,TBST洗膜3次,每次15 min,二抗孵育1 h,加显影液(Millipore 公司),利用X光片(Kodak公司)经过洗片机(102型,Kodak公司)处理后显影。
1.4 细胞超声和染色质免疫共沉淀
细胞超声和染色质免疫共沉淀所需要的试剂及配制方法参考文献[9]配制。取适量细胞,向培养基中加入37%甲醇溶液至终浓度为1%,室温孵育10 min, 加入2 mol/L 甘氨酸至终浓度为0.125 mol/L,室温孵育10 min,终止交联。用冷的PBS 溶液清洗细胞3 遍,加入适量裂解缓冲液1(140 mmol/L NaCl,50 mmol/L HEPES pH 7.9,1 mmol/L EDTA, 0.5% NP‑40, 0.25% Trition‑X100,10%甘油),冰上孵育10 min,离心;加 入 适 量 裂 解 缓 冲 液2 (10 mmol/L Tris‑HCl pH 7.9, 200 mmol/L NaCl, 1 mmol/L EDTA,0.5 mmol/L EGTA),冰上孵育10 min;用裂解缓冲液3 (10 mmol/L Tris‑HCl pH 7.9, 100 mmol/L NaCl,1 mmol/L EDTA,0.5 mmol/L EGTA,0.1%脱氧胆酸钠,0.5%月桂酰基氨基酸)重悬。用Covaris M220超声仪进行细胞超声30 min。取适量染色质加入适量抗体,4℃摇床结合过夜。取适量protein A 或 者protein G Dynabeads (Invitrogen,10002D),用裂解缓冲液(50 mmol/L HEPES pH 7.9,150 mmol/L NaCl,2 mmol/L EDTA,1%Triton X‑100,0.5%脱氧胆酸钠)重悬。4℃摇床孵育1~2 h。依次用高盐缓冲液(50 mmol/L HEPES pH 7.9,500 mmol/L NaCl,2 mmol/L EDTA,1%Triton X‑100,0.5%脱氧胆酸钠)、LiCl 缓冲液(10 mmol/L Tris‑HCl pH 7.9,250 mmol/L LiCl,2 mmol/L EDTA,N0.5%P‑40,0.5%脱氧胆酸钠)、TE 缓冲液(10 mmol/L Tris‑HCl pH 7.9,2 mmol/L EDTA)冲洗Dynabeads,再用洗脱液(0.1%十二烷基磺酸钠,10 mmol/L NaHCO3)重悬,60℃振荡孵育1 h,解交联保存,用KAPA Hyper Prep Kit(KAPA biosystems,KK8504)建ChIP‑seq库。
1.5 DNA文库构建
根据KAPA Hyper Prep Kit 推荐的使用方法[10]分为末端修复和末端加“A”、连接“Y”形接头、片段纯化及片段大小筛选、PCR 扩增、纯化DNA片段、DNA PAGE胶检测文库质量步骤。文库构建完成,公司测序,ChIP‑seq文库采用双端测序获得数据。
1.6 Surveyor 实验
Surveyor 核酸内切酶对于DNA 双链中的碱基错配情况具有很强的特异识别能力,在含有错配的DNA 双链经过Surveyor 核酸酶处理后,经过琼脂糖凝胶电泳呈现出两条大小不同的条带,当基因组被gRNA 在特异位点编辑后,扩增该区域经解链‑退火步骤则会在重新随机退火后新的DNA 双链中形成碱基错配现象,用Surveyor核酸内切酶处理退火后的产物可以对Cas9蛋白的切割效率作出评估。
在Trim28 基因组的第一个外显子上下游600 bp 设 计 引 物SUVRYOR‑F:5'‑cagcagttggcgg‑cgagcgcgtct‑3' 和SUVRYOR‑R: 5'‑tcctctcgcccagg‑tggcatcctcat‑3'。 使 用 常 规PCR 扩 增 方 法 和JumpStartTMTaq Ready Mix(Sigma 公司)分别在WT、gRNA1+gRNA2 pool、gRNA3+gRNA4 pool基因组DNA 样品中扩增目的条带,设计退火程序:-0.3℃/s,从95℃退火至25℃,使用Surveyor®Mutation Detection Kits (Integrated DNA Technologies公司)加入0.5 μl Surveyor Enhancer S和Surveyor Nuclease S,放于42℃水浴锅孵育30 min,利用琼脂糖凝胶电泳评估gRNA 切割效果。
1.7 生物信息学分析
野生型和基因敲除型RNA‑seq样品各有3个生物学重复,生物信息学分析的标准化参数在每个IGV 示意图的左上部分。测序数据经过FastQC[11]检验后,经过Trimmol/Lomatic[12]软件处理去掉接头及低质量序列。RNA‑seq 数据通过Tophat[13]软件比对到人hg38 基因组,ChIP‑seq 数据通过Bowtie2[14]软 件比 对 到 人hg38 基 因 组。Htseq‑count[15]软件用来计算RNA‑seq数据中各样品比对上的序列数目,DESeq2[16]和R用来计算差异表达基因和基因的RPKM(reads kilobases per million reads) 值。差异表达倍数2 倍以上并且padj(p‑adjusted)<0.05 的基因被定义为显著上调或下调的差异基因。ChIP‑seq数据比对后,取唯一比对的序列,并且去掉冗余的序列进行后续的分析。IGV[17]软件用来数据的可视化,MACS2[18]软件进行数据峰值计算。文中热图(Heatmap)及线图通过R软件绘制。
实验中使用到的抗体及货号:TRIM28 抗体(Active Motif,61174)、H3K27ac 抗体(Abcam,ab4729)、 H3K4me1 抗 体(Abcam, ab8895)、H3K4me3抗体(Abcam,ab8580)、H3K9me3抗体(Abcam, ab8898)、 PCDHB14 抗 体(Abgent,A‑AP12321a) 、 TUBULIN 抗 体 (ABclonal,AC008)、ZNF93抗体(Abgent,A‑AP20006A)。
2 结 果
2.1 成功构建Trim28基因敲除细胞系和TRIM28回补细胞系
利用CRISPR/Cas9 技术,设计靶向Trim28 起始密码子下游第一位外显子分别切割不同位置的gRNA1、gRNA2、gRNA3 和gRNA4。将4 种不同的gRNA 分为两组,gRNA1 和gRNA2 为一组,gRNA3和gRNA4为一组,两组HEK293F细胞系分别转染两组gRNA 质粒(图1a),分别提取野生型HEK293F细胞系、gRNA1+gRNA2 pool和gRNA3+gRNA4 pool 细胞系的基因组DNA,Surveyor 实验检测gRNA 切割效率。根据琼脂糖凝胶电泳结果,与野生型HEK293F 细胞系比较,发现gRNA1+gRNA2 pool 和gRNA3+gRNA4 pool 细胞系DNA 条带底部具有箭头所示的弥散状条带(图1b),证明Cas9‑gRNA切割DNA片段导致基因组DNA碱基产生错配。96 孔板分单克隆,利用SUVRYOR‑F 和SUVRYOR‑R 引物进行常规PCR,PCR 产物进行Sanger 测序,测序结果和野生型对比,获得两株Trim28基因组产生移码突变的细胞系,其中一株碱基缺失193 bp,称为TRIM28 KO1细胞系,另一株碱基缺失412 bp,称为TRIM28 KO2 细胞系(图1c)。利用免疫印迹技术对比野生型HEK293 细胞系TRIM28蛋白表达量,结果证明在Trim28基因缺失的细胞系中Trim28 完全敲除,蛋白质表达显著降低(图1d)。通过构建Trim28 cDNA 质粒在Trim28 基因敲除细胞系中回补Trim28 基因,免疫印迹结果证明回补的TRIM28 蛋白量和野生型HEK293F细胞系TRIM28蛋白表达量基本一致(图1e),这些结果表明Trim28 基因敲除细胞系和TRIM28回补细胞系被成功构建。

Fig.1 Generation of Trim28 gene knockout cell line and TRIM28 rescue cell line(a) Schematic representation of the knockout targeting strategy, the targeting region located in exon 1. (b) Surveyor assay result shows Cas9 gRNA efficiency.Arrows at the bottom of main band indicate the smear band reflecting the efficiency of Cas9 gRNA.(c)Trim28 genomic DNA was deleted in Trim28 KO1 and Trim28 KO2 cell lines. (d) Knockout efficiency was detected by Western blot in HEK293 cell line. (e) Rescue efficiency was detected by Western blot.
2.2 TRIM28抑制内源表达量较低的基因的转录
为了深入研究TRIM28 的功能,本文使用mRNA‑seq 测序技术分析野生型HEK293F 细胞系和Trim28 基因敲除细胞系中转录组的变化。根据结果发现,和野生型HEK293F 细胞系相比,在Trim28基因敲除细胞系中大部分基因的转录水平上升(图2a)。将HEK293F 野生型细胞系和Trim28基因敲除细胞系中基因的RPKM 值取对数处理后分别分为3 个小组,其中All WT 和All KO 表示在野生型细胞系和Trim28 基因敲除细胞系中所有基因的表达水平,Down KO 和Down WT 表示在Trim28基因敲除细胞系中转录水平下降的那些基因和它们在野生型中的表达水平,Up KO 和Up WT表示在Trim28 基因敲除细胞系中转录水平上升的那些基因和它们在野生型中的表达水平。将Up KO和Up WT组进行比较,发现在Trim28基因敲除细胞系中转录水平上升的那些基因在野生型属于低表达基因(图2b),这些结果说明TRIM28 主要抑制内源表达水平较低的基因转录。

Fig.2 Up regulated genes by Trim28 KO are low expressed genes in HEK293F cells(a)The heatmap shows the up and down genes in Trim28 KO cell line.(b)Box plot shows the up regulated genes in WT(Up WT)are low expressed genes compared to other groups.
2.3 TRIM28抑制锌指蛋白和原钙黏蛋白基因的转录
为了进一步研究TRIM28的功能,将Trim28基因敲除细胞系中转录水平上升的基因进行分类,根据GO 分类结果发现,TRIM28 主要抑制两类基因的转录,一类是锌指蛋白(ZNF)基因,另一类是原钙黏蛋白β(PCDHβ)基因(图3a)。锌指蛋白指的是由一类含有大约30 个氨基酸的环和一个与环上的4 个半胱氨酸或2 个半胱氨酸和2 个组氨酸配位的锌离子构成的结构像手指状的蛋白质,锌指蛋白通过和DNA或者RNA相互作用在生物体内参与细胞分化、信号传导、细胞迁移等多种生物学功能[19]。在锌指蛋白家族中,有一类N 端由KRAB(Kruppel‑associated box) 结 构 域组 成 的 锌 指 蛋白[20]。既往报道发现,TRIM28在B细胞中能够抑制含有KRAB 结构域的锌指蛋白转录[21],这和本文的实验结果一致(图3a)。
原钙黏蛋白分为α、β 和γ 3 个亚单元,根据mRNA‑seq 和GO 分 析 结果(图3a,b) 发现,TRIM28 能够抑制原钙黏蛋白β 基因的转录,敲除Trim28基因后转录水平显著升高的主要是β家族基因。原钙黏蛋白在神经细胞中高表达,是一种依赖钙离子的黏附分子,分布于神经细胞膜表面,主要是参与信号传导作用,帮助神经细胞参与识别“自我”和“非我”功能[22]。原钙黏蛋白群在大脑神经网络发育中起到重要作用,若通过基因敲除或下调原钙黏蛋白,可导致神经细胞发育异常、彼此不分,并诱发凋亡[4]。既往研究集中在DNA 甲基化和染色质高级结构与原钙黏蛋白转录调控之间的关系[6,23],这是首次发现TRIM28 和PCDHβ 家族转录调控之间的联系。
2.4 在Trim28基因敲除细胞系中锌指蛋白家族和原钙黏蛋白家族的蛋白质水平升高
本文发现在Trim28 基因敲除细胞系中部分锌指蛋白和原钙黏蛋白β的转录水平显著上升,选择锌指蛋白ZNF93 和原钙黏蛋白PCDHβ14 这两个基因作为代表,检测它们的蛋白质表达水平是否发生变化。免疫印迹(图4a)的结果表明,和野生型HEK293F细胞系中ZNF93和PCDHβ14的蛋白质表达水平相比,在Trim28 基因敲除细胞系中这两种蛋白质的表达水平显著上升。由于mRNA‑seq数据的RPKM 值(图4b)在一定程度上能够发映出mRNA水平,因此认为TRIM28抑制锌指蛋白和原钙黏蛋白的转录活性。当Trim28 基因敲除后,这两类基因的转录水平显著上升,蛋白质表达水平和野生型相比显著升高。

Fig.3 TRIM28 mainly suppresses the transcription of ZNF family genes and PCDHβ family genes(a)GO analysis shows up‑regulated genes.(b)RNA‑seq signal shows the increased of PCDHβ family genes in Trim28 KO cell line.
2.5 Trim28基因敲除导致基因组组蛋白修饰发生改变
已经证明TRIM28抑制基因的转录,在Trim28基因敲除细胞系中转录水平上升的基因数量大于转录水平下降的基因。为了更深入地研究TRIM28与组蛋白修饰的染色质高级结构之间的关系,本文进行了H3K4me1修饰、H3K4me3修饰、H3K9me3修饰、H3K27ac 修饰、RNA Pol II 和TRIM28 的染色质免疫共沉淀实验。根据实验结果(图5b)发现,TRIM28 主要富集在有H3K9me3 修饰、H3K4me3修饰和H3K27ac 修饰的位点上。H3K9me3 修饰是异染色质的标志,和基因沉默具有相关性,H3K4me3 修饰和H3K27ac 修饰是启动子与增强子的标志物[24],这说明TRIM28 不仅参与异染色质的建立,还通过远端调控元件调控基因的转录。根据这些修饰在野生型细胞系和Trim28 基因敲除细胞系中的变化(图5a),发现TRIM28 主要参与异染色质的建立过程,敲除Trim28 导致全基因组范围的H3K9me3修饰大幅度降低,H3K4me1修饰和H3K4me3 修饰只有部分降低。根据实验结果,认为TRIM28 不仅参与其位点的H3K9me3 修饰的建立也会参与H3K4me3修饰的建立。
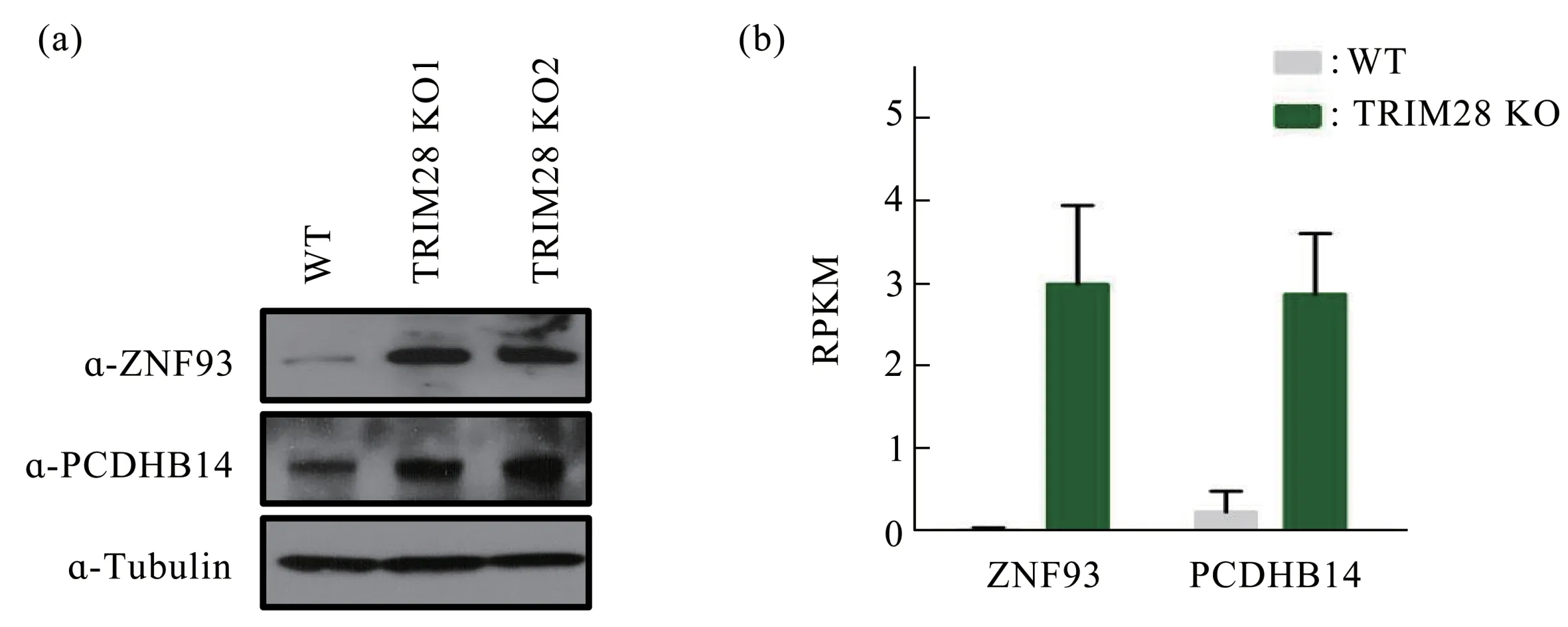
Fig.4 The protein levels of ZNF and PCDHB increased in Trim28 knockout cell lines(a) Protein level was detected by Western blot in HEK293 cell line and Trim28 KO cell line. (b) RPKM of ZNF93 and PCDHβ14 are shown in WT and Trim28 KO cell line.
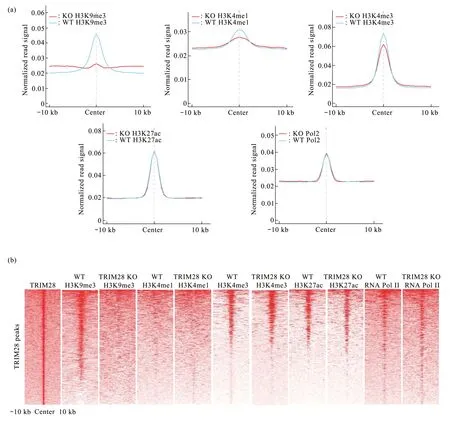
Fig.5 TRIM28 KO-mediated histone changes at TRIM28 binding sites(a) Line plots show the histone modification enrichment at TRIM28 binding sites. (b) The heatmap shows the histone modification enrichment at TRIM28 binding sites.
2.6 Trim28基因敲除导致锌指蛋白和原钙黏蛋白基因组蛋白修饰发生改变
为了更深入地研究TRIM28抑制锌指蛋白和原钙黏蛋白的机制,本文分析了这两类基因上组蛋白修饰的变化,发现H3K9me3 修饰和野生型HEK293F 相比大幅度降低,在锌指蛋白家族基因上H3K4me1、H3K4me3 和H3K27ac 都 大幅 度升高,这说明染色质的状态由致密变得松散,还发现锌指蛋白是TRIM28的直接靶基因。根据实验结果(图6a)认为,TRIM28 通过参与锌指蛋白基因区域H3K9me3 修饰的建立抑制其转录活性,当敲除Trim28 后H3K9me3 修饰降低,染色质状态由致密变得松散,提高了锌指蛋白的转录活性。本文发现在PCDHβ 基因上,H3K9me3 修饰大幅度减少,H3K4me3 修饰显著上升, H3K4me1 修饰和H3K27ac修饰没有明显的变化(图6b)。之前对于原钙黏蛋白基因的研究集中在染色质构架蛋白CTCF通过影响染色质高级结构激活特定的原钙黏蛋白启动子调控原钙黏蛋白的转录[15‑16],本文认为,敲除Trim28 导致染色质结构松散,染色质高级结构发生改变,原钙黏蛋白β的转录活性也随之上升。
2.7 在Trim28敲除细胞系中回补Trim28会部分抑制锌指蛋白和原钙黏蛋白的转录活性
本文在Trim28 基因敲除细胞系中回补了Trim28,认为在Trim28 基因敲除细胞系中染色质的结构已经发生了改变,染色质结构的变化会导致一些染色质相关因子的定位发生变化,即使回补Trim28 基因,染色质相关因子的定位可能不会完全恢复,因此在Trim28 回补的细胞系中只能部分抑制锌指蛋白和原钙黏蛋白的转录(图7)。
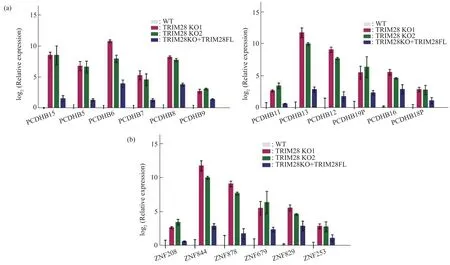
Fig.7 Reintroduction of TRIM28 full length in Trim28 KO cell line could partially suppress the expression of ZNF family and PCDHβ familyRT‑qPCR of PCDHβ and ZNF genes in WT,Trim28 KO and TRIM28 rescue(TRIM28KO+TRIM28FL)cell lines.
3 讨 论
TRIM28 最早被鉴定能够抑制锌指蛋白的转录[1],随后的研究发现TRIM28通过和异染色质相关蛋白HP1、SETDB1相互作用,在靶基因上建立H3K9me3 修 饰 从 而 抑 制 基 因 的 转 录[17‑18]。对 于TRIM28的其他生物学功能并不清楚,为了更深入地研究TRIM28的功能,本文首先建立了Trim28基因敲除细胞系,通过RNA‑seq发现TRIM28主要是抑制内源表达较低基因的转录。然后,通过GO分析发现,TRIM28能够抑制两类基因的转录,锌指蛋白基因和原钙黏蛋白β基因。为了进一步解释其中的机理,本文又进行了染色质免疫共沉淀实验,发现组蛋白修饰和TRIM28之间的关系,在Trim28基因敲除细胞系中,H3K9me3 修饰下降,导致染色质结构松散,使得基因的转录活性增强。在锌指蛋白基因区域,TRIM28 直接参与异染色质的建立,维持基因的沉默。TRIM28通过间接作用调控原钙黏蛋白基因的转录活性。
既往对于原钙黏蛋白基因的研究集中在,染色质构架蛋白CTCF通过影响染色质高级结构,激活特定的原钙黏蛋白启动子,调控原钙黏蛋白的转录[25‑26]。根据实验结果推测,在Trim28 基因敲除细胞系中染色质高级结构的改变导致染色质相关因子CTCF在原钙黏蛋白区域的分布发生变化,从而引起启动子区域H3K4me3 修饰的变化,增强原钙黏蛋白的转录活性。
4 结 论
本文利用CRISPR/Cas9 技术,通过在HEK293 F细胞系中建立Trim28基因敲除型细胞作为实验材料,通过综合运用生物信息学和蛋白质组学的方法,鉴定出TRIM28抑制锌指蛋白家族基因和原钙黏蛋白家族基因,初步探讨了组蛋白修饰和TRIM28 转录抑制基因之间的关系,发现TRIM28主要是通过在基因组范围内建立H3K9me3 修饰抑制基因的转录,为更深入地研究TRIM28和染色质高级结构以及原钙黏蛋白家族基因之间的分子机制提供了新的思路。

