一种预测个体肿瘤的抗癌药物反应分类计算模型及其应用*
李少达 李玉双
(燕山大学理学院,秦皇岛 066004)
癌症的异质性和遗传多样性,导致同种癌症的患者即使采用相同的治疗方法,也有可能得到不同的疗效[1‑3]。从患者的角度,更希望了解给定药物是否有效。针对特定癌症类型,如何在分子水平上探索癌细胞系对抗癌药物的反应已成为精准医疗的研究热点之一[4]。基因组学的快速发展及人类基因组计划的顺利实施,诞生了海量的生物学数据,为在分子水平上预测抗癌药物临床反应提供了良好的数据基础[5]。特别值得一提的是,2012年《自然》(Nature)杂志发表了两项系统的大规模研究,癌症基因组计划(CGP)[6]和癌细胞百科全书(CCLE)[7],研究中所涉及到的细胞系几乎涵盖所有常见的癌症类型,使得完全以数据驱动、计算建模的方式自动识别生物标志物,系统解析抗癌药物反应与癌症细胞系基因谱之间的关系成为可能。
研究人员借助这些数据集开发抗癌药物反应预测计算模型[8‑11]的主要思想有两种,一种是基于核方法预测药物敏感性,其中最具有代表性的模型之一为支持向量机(SVM)。如Hejase 等[12]基于美国国家癌症研究中心(NCI)数据,应用非线性SVM成功预测了药物化合物对乳腺癌细胞的影响;Wang等[13]基于CCLE数据集,利用基因突变、拷贝数变异和基因表达等数据,通过组合SVM 模型对三类特定组织下的细胞系进行了敏感性分类。另一种是基于特征提取方法预测抗癌药物反应。如最近Su等[14]融合基因表达和拷贝数变异,构建了两类 深 度 反 应 森 林(Deep‑Resp‑Forest) 模 型MIMGS1 和MIMGS2,成功预测抗癌药物对细胞系的敏感或抑制。
上述模型大大推进了抗癌药物反应预测的研究进程,但在模型预测性能、应用范围等方面仍有可探索的空间。受以上工作启发,本文聚焦抗癌药物敏感‑抑制二分类问题,构建了mRMR‑SVM模型,从细胞系的基因表达数据出发,利用“最大相关最小冗余”算法[15](mRMR)提取特征基因,借助SVM 进行分类预测,不仅降低了时间运行成本,而且提升了模型的预测性能和生物可解释性。
1 数据来源和数据处理
从CCLE数据库(http://www.broadinstitute.org/ccle)下载了1 036 个癌症细胞系的53 619 个基因表达信息,以及504个细胞系对24种药物的敏感性数据(敏感性指标为activity area)。进一步选出462 个既有基因表达又有药物敏感性数据的细胞系,并用z‑score 方法标准化敏感性数据。依据文献[14],如果细胞系对药物的敏感性值(标准化后的敏感性值)大于0.8,定义为“细胞系对药物是敏感的”,如果小于-0.8,定义为“细胞系对药物是抑制的”,其余数据定义为冗余数据,不参与实验。在此定义下,有2 种药物对应的细胞系很少,故在实验中舍去,保留其余22 种药物进行分类预测,其对应细胞系的数量范围是93~215。
为验证模型的泛化性能,选取另一数据库进行实验。从癌症药物敏感性基因组学数据集(GDSC)(https://www.cancerrxgene.org) 下 载 了789 个癌细胞系的12 072 个基因表达信息,655 个癌细胞系对140种药物的敏感性数据(敏感性指标为IC50)。采用与CCLE 相同的处理方式,选取11种药物进行分类预测,其对应细胞系的数量范围是76~179。
2 mRMR-SVM模型
本文首先利用mRMR 提取特征基因,然后构建SVM预测抗癌药物反应分类及识别生物标志物。具体流程如图1所示。
2.1 特征基因的选取

Fig.1 The flowchart of mRMR-SVM
由于CCLE的基因数量大,许多基因的表达值差别不明显,为降低模型运行成本,本文先计算每个基因在所有细胞系下的表达方差,再将基因按方差从高到低的顺序进行排序,选取表达差异较大的前10 000 个基因作为候选特征基因。然后利用mRMR 算法提取得分最高的基因集合作为最终的特征基因集。具体定义如下:设x,y为随机变量,p(x),p(y),p(x,y)为概率密度函数,则x 和y之 间 的 互 信 息 为 : I(x; y) =设S为基因表达向量的集合(为方便计算,本文选取|S|= 500),c为给定药物在所有细胞系下观测到的“敏感‑抑制”类别向量:如果第j 个细胞系对给定药物是敏感的,其分量cj= 1;如果第j 个细胞系对给定药物是抑制的,cj=-1。定义S与c的相关度D(S,c)=即S 中的基因表达向量与类别向量c 之间的所有互信息值的平均值,这里xi为S中第i 个基因在所有细胞系下的表达向量。定义S的冗余度即S 中基因表达向量之间所有互信息值的平均值。定义S的得分
由于GDSC的基因数量相对较小,故在实验中不需进行基因初筛,直接利用mRMR 算法提取特征基因。
2.2 mRMR-SVM的构建
SVM 解决线性不可分问题的主要思想是:将原始低维线性不可分的分类空间映射到高维的特征空间,只要映射的空间维数足够高,则原始空间将转换为一个新的线性可分空间。通过在线性可分空间建立一个最优的决策超平面,使得距离分类平面两侧最近的训练样本之间距离最大,将线性不可分的数据转化为线性可分。本文构建的SVM 包含两个参数,即低维空间映射到高维空间的核函数以及惩罚因子C。
利用mRMR 选取的500 个特征基因的表达数据和观测到的反应分类标签来训练SVM,具体的mRMR‑SVM构建过程如图2所示。
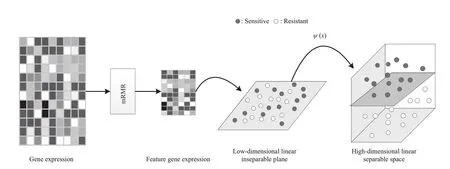
Fig.2 The construction of mRMR-SVM
采用交叉验证确定模型参数及评估模型性能。首先,将数据集随机分为90%的训练集和10%的测试集;为防止模型过拟合,再将训练集随机平均划分为5 份,分别用其中4 份作为训练集,1 份作为验证集训练模型超参数;最后,用测试集进行模型性能评估。上述过程重复执行5次,测试集分类结果的均值为最终预测结果。本文所用编程语言为Python,代码见本文网络版附件。
2.3 模型的评价指标
本文采用ACC、AUC、precision、recall 和F1 5个指标来评价模型的预测性能。
ACC为模型的预测准确率,具体定义为:
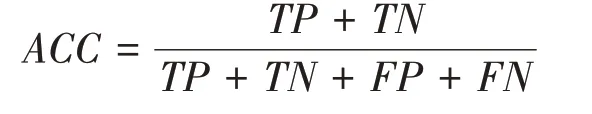
其中,TP 为预测是敏感而实际也是敏感的细胞系数量,TN为预测是抑制而实际也是抑制的细胞系数量,FP 和FN 为预测是敏感和抑制而实际则相反的细胞系数量。
AUC(area under curve)为利用预测结果所绘制的ROC 曲线下面积,ROC 曲线的纵坐标为TPR(true positive rate),横坐标为FPR(false positive rate),这里TPR为真阳率,FPR为假阳率。
precision为精确率,反应了模型预测为敏感的细胞系的预测准确率,定义为:
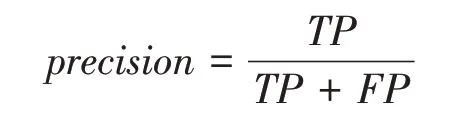
recall 为召回率,反应了模型对敏感细胞系的预测准确率,定义为:
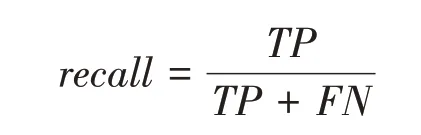
F1 得分是综合precision 和recall 给出的平均定义,其值越大,说明模型预测性能越好。定义为:
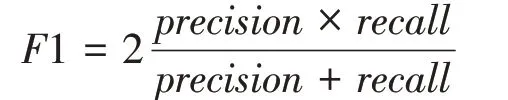
3 抗癌药物反应分类预测结果
模型训练的最优核函数为linear。对于惩罚因子C,本文采用了两种确定方法,第一种直接使用模型默认的参数1,第二种针对每种药物单独调整选出最优参数。
3.1 基于CCLE数据集的预测结果分析与比较
针对CCLE数据集的22种药物,选取C为默认值1的mRMR‑SVM预测结果(表1):22种药物的平 均ACC、AUC、precision、recall、F1 分 别 为0.897、0.966、0.898、0.892、0.888。单独调C 的预测结果(表2):平均ACC、AUC、precision、recall、F1 分 别 为0.904、0.969、0.905、0.898、0.895。从预测结果可以看出,单独调C 的模型预测结果更理想。
为了阐释mRMR 算法提取的500 个特征基因对抗癌药物反应分类预测的影响,一方面,利用mRMR算法提取的500个特征基因训练深度神经网络(DNN),简称mRMR‑DNN。使用网格搜索法调参,最终确定mRMR‑DNN 包含3 个隐藏层,每个隐藏层的神经元个数分别为60、30 和30,层与层之间的激活函数分别为tanh、rectifier 和linear,输出层的激活函数为softmax。另一方面,从经过方差筛选出的10 000 个基因中随机挑选500 个基因,训练SVM 和随机森林(RF),两种模型均采用网格搜索法调参,SVM 的最终参数C=0.1,RF的参数(决策树的个数)为80。
Table 1 Classification result of mRMR-SVM on CCLE data set(C=1)

Table 2 Classification result of mRMR-SVM on CCLE data set(C separately adjusted)
在共同讨论的14种抗癌药物反应分类预测中,mRMR‑SVM(单独调参)的平均ACC 为0.911,mRMR‑DNN为0.899,SVM为0.525,RF为0.526,文献[14]中的MIMGS1 和MIMGS2 分别为0.858、0.850,说明mRMR 算法提取的500 个特征基因对抗癌药物反应分类预测至关重要。图3从整体上展示了以上6 种模型的预测性能,mRMR‑SVM 明显优于其他5种模型。此外,与文献[16]中的CDCN模型进行比较,在共同讨论的22 种抗癌药物的反应分类预测中,mRMR‑SVM的平均ACC为0.904,高于CDCN(0.566)。
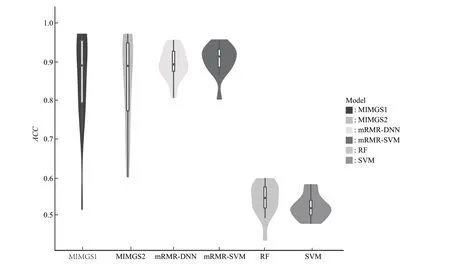
Fig.3 The classification accuracy of six models on CCLE data set
3.2 基于GDSC数据集的预测结果分析与比较
针对GDSC数据集中的11种药物,表3展示了mRMR‑SVM 在C 为默认值1 时的预测结果:平均ACC、AUC、precision、recall、F1 分别为0.839、0.909、0.850、0.840、0.834。单独调C的预测性能进一步提升(表4),平均ACC、AUC、precision、recall、F1 分 别 为0.851、0.917、0.865、0.848、0.845。
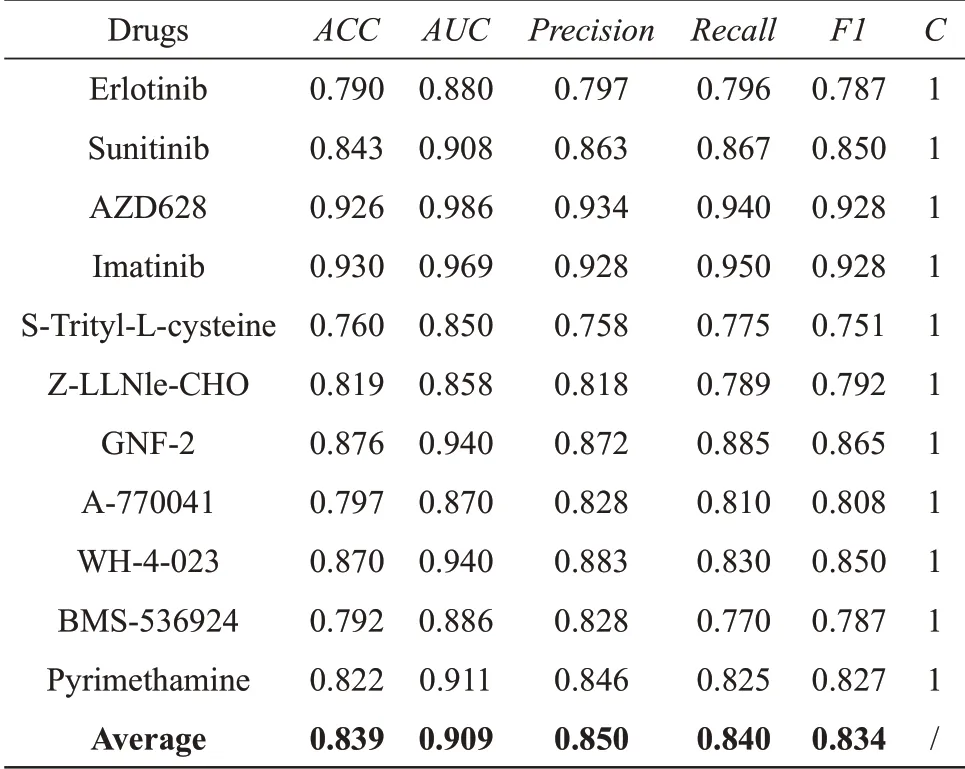
Table 3 Classification result of mRMR-SVM on GDSC data set(C=1)
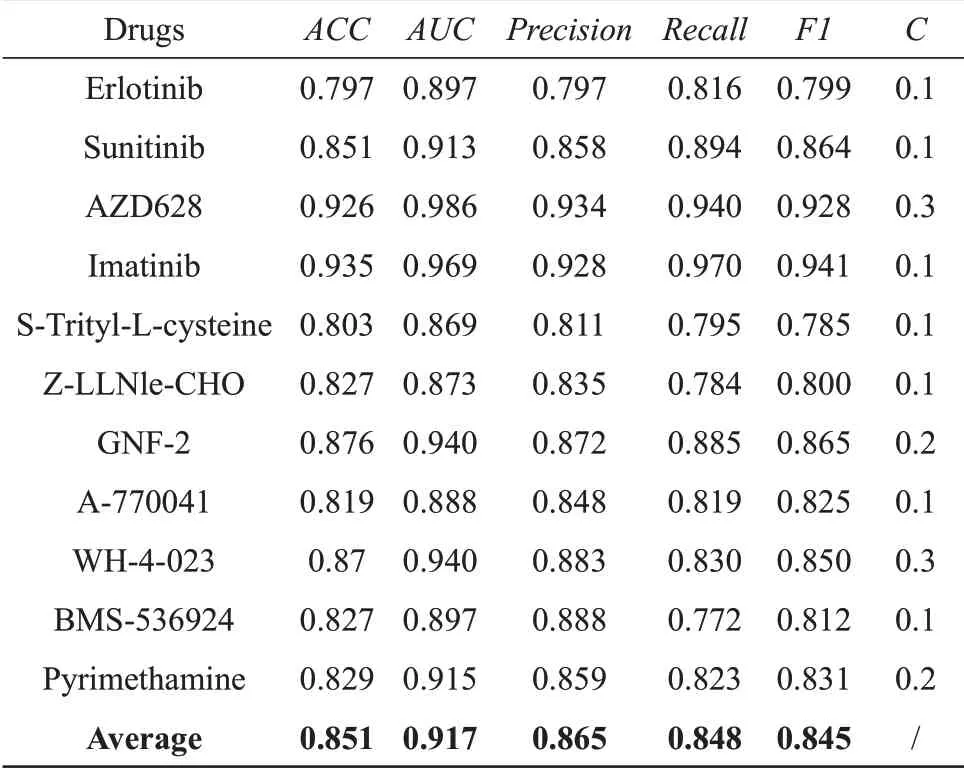
Table 4 Classification result of mRMR-SVM on GDSC data set(C separately adjusted)
对于GDSC 数据集,本文同样训练了mRMR‑DNN:包含3个隐藏层,每层的神经元个数分别为63、78 和86,层与层之间的激活函数分别为rectifier、linear 和tanh,输出层的激活函数为softmax。同样地,从GDSC 数据集12 072 个基因中随机挑选500 个作为特征基因,训练了SVM(C=0.9)和RF(决策树个数为95)。针对共同讨论的11 种药物,mRMR‑SVM(单独调C)的平均ACC 为0.851, mRMR‑DNN 为0.817, SVM 为0.652,RF 为0.640,MIMGS1 为0.805,MIMGS2为0.815。图4 从整体上展示了6 种模型的预测结果,mRMR‑SVM 的预测性能明显优于其他5 种模型。此外,针对共同讨论的7 种药物,mRMR‑SVM的平均ACC为0.861,高于文献[16]的CDCN模型(0.630)。
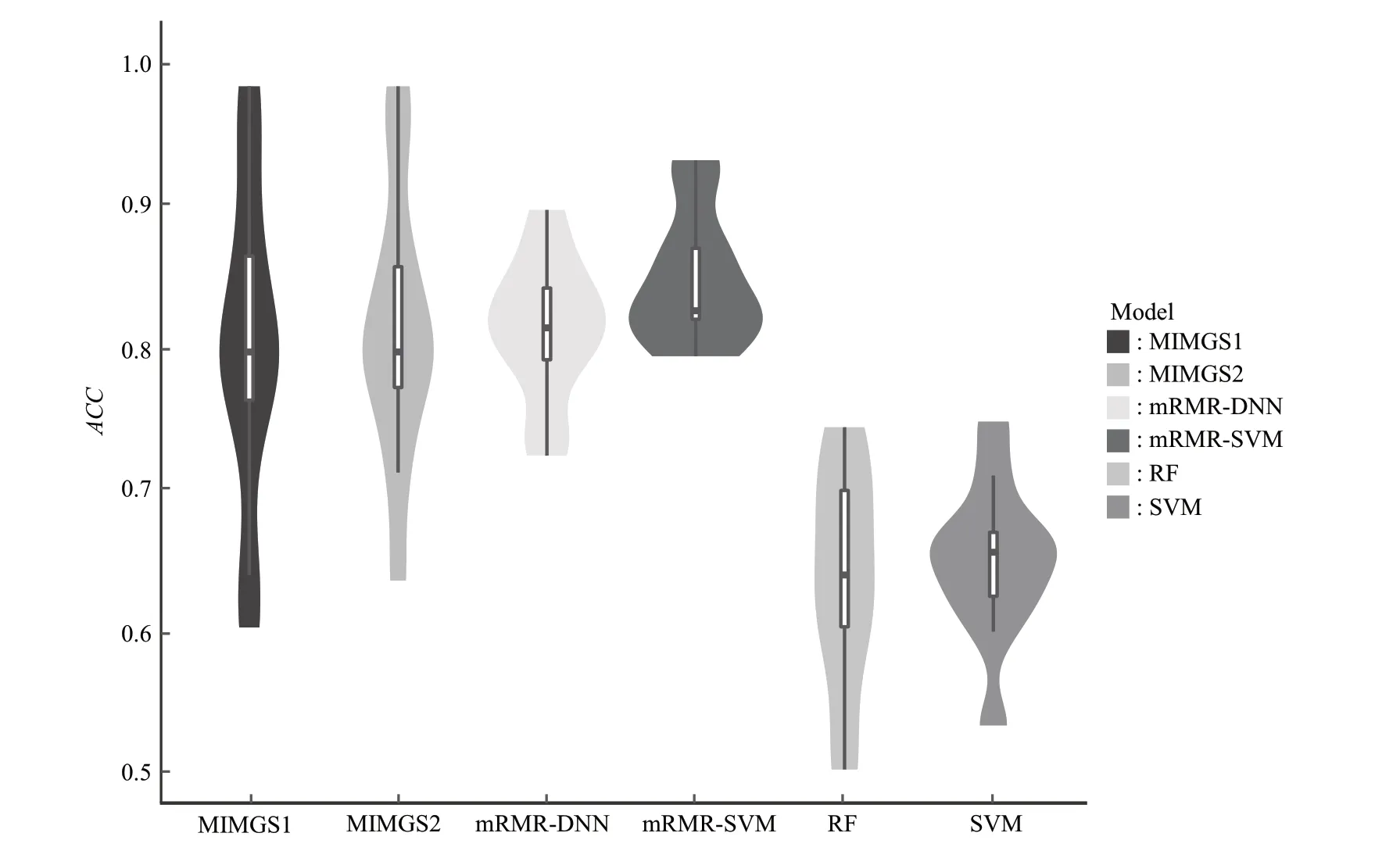
Fig.4 The classification accuracy of six models on GDSC data set
3.3 基于三类特定组织的预测结果分析与比较
为进一步验证mRMR‑SVM的泛化能力,受文献[13]的启发,对CCLE数据集中三类特定组织下的细胞系,包括造血和淋巴组织(包含71 个细胞系)、皮肤组织(包含40 个细胞系)、肺组织(包含94个细胞系),针对22种抗癌药物进行反应分类预测。考虑到模型的泛化性,参数C 取默认值1。五次五折交叉验证得到的平均预测结果如表5 所示。三类特定组织的平均AUC 依次达到了0.973、0.981、0.965,均优于文献[13]中基于基因表达、拷贝数变异、基因突变等多类数据融合的SVM(其平均AUC 依次为0.81、0.82、0.83)。该实验表明,mRMR‑SVM 对于小样本数据集同样具有很好的预测能力。
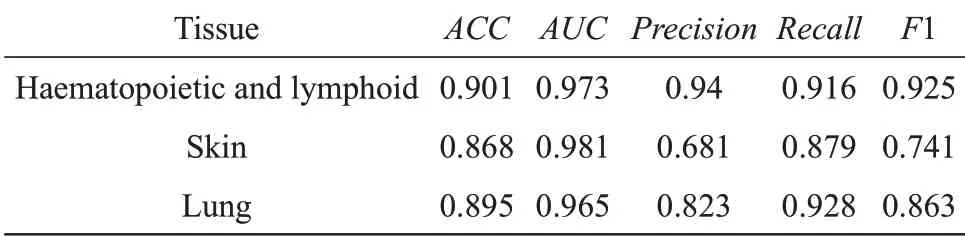
Table 5 Average classification result of mRMR-SVM on three kinds of tissues
4 生物标志物的识别
mRMR‑SVM 能够识别出许多与癌症发生、发展密切相关的重要基因,为抗癌药物生物标志物的筛选提供理论参考。如在抗癌药物17‑AAG的特征基因中排序第二的TP73‑AS1,已被证实在大多数肿瘤中高表达,在乳腺癌、胃癌和肝癌等肿瘤中发挥促癌基因作用,在膀胱癌中低表达并发挥抑癌基因作用[17]。PARK7在4种药物17‑AAG、Nutlin‑3、Panobinostat和RAF265中均被选为Top基因。事实上,PARK7 已被确定为各种癌症的发病机制和生存的高危因素,它增强了肿瘤的起始、增殖、转移和复发,以及对化疗的抵抗力[18]。有文献发现,PQLC2 的上调对于体外和体内胃癌的发展至关重要,靶向PQLC2是胃癌治疗的有效策略[19]。本文验证了PQLC2 不仅是具有抗胃癌活性药物ZD‑6474 的Top 基因,而且在另外3 种抗癌药物PD‑0332991、TAE684和Topotecan的特征基因排序中也位居前5。IFI6 是一个能被Ⅰ型干扰素诱导上调的干扰素刺激基因,在多种恶性肿瘤中高表达,能够抵抗细胞凋亡,对肿瘤的放化疗效果有一定影响[20]。文中IFI6 在5 种药物17‑AAG、Erlotinib、TKI258、ZD‑6474 和AZD0530 的特征基因排序中均位居前10,与已有结果一致。
5 结 论
本文提出的mRMR‑SVM 不仅在公共数据集CCLE、GDSC,以及三类特定组织中取得了较好的分类预测结果,而且能够识别与抗癌药物反应相关联的生物标志物,说明其可以作为抗癌药物反应分类预测的有效工具。此外,mRMR‑SVM 具有可拓展性,可融入其他类型数据(如基因突变等)进一步提升模型的预测性能。对于模型筛选的特征基因,可以构建特征基因信息与药物敏感性之间的回归模型(如岭回归、逻辑回归),通过回归系数挖掘抗癌药物敏感性预测因子。
附件 PIBB_20210082‑prgm‑S1.zip 请见本文网络版(www.pibb.ac.cn或www.cnki.net)。

