基于ITS 和ISSR 的灵芝种质资源遗传多样性分析
许 琳,张 瑞,王 胜,李金涛,刘琳玲,闫梅霞
(1.中国农业科学院 特产研究所,吉林 长春 130112;2.吉林农业大学 中药材学院,吉林 长春 130118)
灵芝(Ganoderma lucidum)是担子菌纲多孔菌科灵芝属真菌,为我国传统名贵药材,被称为“仙草”,在我国已有上千年的栽培及应用历史。我国是世界上最大的灵芝生产国和消费国,2020 年灵芝和灵芝孢子粉合计年产量约1.7 万t,产业总产值超过80 亿元[1]。灵芝子实体开展药食同源试点管理[2]、破壁灵芝孢子粉保健食品实施备案制[3]等利好政策的实施将进一步促进灵芝产业健康发展。目前,我国灵芝主栽区包括武夷山及龙泉、东北吉林长白山、安徽大别山、山东鲁西四大区域[4]。吉林作为灵芝主要栽培区之一,主要栽培品种为赤芝、松杉灵芝、韩芝等[5],存在灵芝种源引进随意、来源不清晰的问题,不利于灵芝良种选育工作的开展。因此,有关栽培菌株的鉴定及遗传多样性分析的研究亟需提上日程。
核糖体DNA 内转录间隔区(Ribosomal DNA internal transcribed spacer,rDNA-ITS)基因是目前真菌属内种间鉴定可靠的基因片段[6],在物种和亚种水平上的识别信息最为丰富[7]。研究表明,ITS 对于灵芝多数种类来说是可靠的区分方法,我国具有ITS 分子序列的灵芝种类有39 种[8],因此,近年来被广泛应用于灵芝种质资源的鉴定及评价[9-10]。拮抗试验操作简单且成本低,常用于菌株的初步鉴定及分类[11],也可有效反映遗传多样性[12-13]。简单序列重复间隔区(Inter-simple sequence repeat,ISSR)标记是对2 个彼此相近、方向相反的简单重复序列(Simple sequence repeat,SSR)之间DNA 区域进行扩增[14],其模板质量要求低,具有很好的稳定性和多态性,常用于菌株的筛选、鉴定及遗传多样性分析[15]等。目前,在对食用菌进行鉴定及遗传多样性分析时,常采用上述拮抗试验、ITS 序列分析与分子标记手段中的2 种或3 种方法相结合进行,其结果也更加准确可靠[16-19]。
目前尚未见关于吉林地区灵芝遗传多样性研究的报道,鉴于此,拟通过拮抗试验结合ITS 序列对供试菌株进行鉴定,构建系统发育树,并利用ISSR分子标记构建遗传聚类图谱,对吉林主栽的18种灵芝菌株进行遗传多样性分析,以期为吉林栽培灵芝的遗传育种及生产利用提供依据。
1 材料和方法
1.1 供试材料
18 个灵芝供试菌株由吉林省5 个灵芝产区所取子实体分离纯化所得,菌株现由中国农业科学院特产研究所食药用菌团队保存,灵芝菌株编号见表1。

表1 供试菌株Tab.1 Tested strains
1.2 试剂与仪器
主要试剂:Ezup 真菌基因组DNA 抽提试剂盒(上海生工生物工程股份有限公司)、ITS 引物及ISSR 引物(上海生工生物工程股份有限公司)、2×TaqPCR Master Mix(上海生工生物工程股份有限公司)、DL2000 DNA Marker(日本宝生物工程株式会社)、50×TAE 缓冲液(北京博奥拓达科技有限公司)。
主要仪器:D3024R 高速冷冻离心机(美国赛洛捷克公司)、T100 PCR 仪(美国伯乐公司)、JW300C电泳仪(北京君意东方电泳设备有限公司)、凝胶成像分析系统(美国伯乐公司)、OSE-260 超微量紫外可见分光光度计(天根生化科技有限公司)。
1.3 培养基配制
马铃薯葡萄糖固体培养基(PDA):马铃薯200 g(浸汁)、葡萄糖20 g、磷酸二氢钾1.5 g、磷酸氢二钾1.5 g、无水硫酸镁2 g、琼脂20 g,加水定容至1 L,pH值自然。
1.4 菌丝培养与DNA提取
将活化的18 种灵芝菌株接种到铺有玻璃纸的PDA培养基平皿上,3次重复,25 ℃避光培养。待菌丝长满平皿后刮取菌丝,按照试剂盒方法逐一提取DNA,并检测DNA质量,A260/A280值为1.8~2.0则质量合格,置于-20 ℃冰箱保存备用。
1.5 ITS扩增
选 择 真 菌 通 用 引 物 ITS1 (5′-TCCGTAGGTGAACCTGCGG-3′ ) 和 ITS4 (5′-TCCTCCGCTTATTGATATGG-3′)对样品DNA 进行扩增。反应体系为25 μL,包含2×PCR Mix 12.5 μL、上下游引物各1.25 μL、DNA 模板1.25 μL、ddH2O 8.75 μL。PCR扩增条件:94 ℃预变性3 min;94 ℃变性30 s、54 ℃退火30 s、72 ℃延伸45 s,30 个循环;72 ℃延伸10 min,终止温度为4 ℃。配制1%的琼脂糖凝胶、1×TAE 电极缓冲液,上样量3 μL,120 V 电压恒流30 min,检测是否有目的条带。
1.6 拮抗试验
18 种灵芝菌株在PDA 培养基活化后,采用陈强[20]等的方法使用直径6 mm 的打孔器打取各菌株种块,接种于直径90 mm 的培养皿内25 ℃对峙培养,每个菌株至少3 次重复。菌丝接触3 d 后,观察记录菌株两两之间的拮抗情况。
1.7 ISSR引物筛选及扩增
参照徐凯[21]、邢志伟[22]、RAMAN 等[23]的方法,对20 个常用ISSR 引物进行分析,筛选出12 条扩增条带清晰、多态性强、稳定性好的引物,同时筛选各引物适宜的退火温度。筛选的引物序列及确定的适宜退火温度见表2。

表2 ISSR引物参数Tab.2 ISSR primer parameters
反应体系为20 μL,包含2×PCR Mix 10 μL、引物1 μL、DNA 模板1 μL、ddH2O 8 μL。PCR 扩增条件:94 ℃预变性3 min;94 ℃变性1 min、退火1 min、72 ℃延伸1 min,40 个循环;72 ℃延伸5 min,终止温度为4 ℃。配制1.5%的琼脂糖凝胶、1×TAE 电极缓冲液,上样量5 μL,以DL 2000 Marker 为指示标量,120 V 电压恒流1 h,于凝胶成像系统上检测并拍照记录。
1.8 数据处理
将PCR 扩增产物交由上海生工生物工程股份有限公司进行测序,所测得的ITS 序列使用SnapGene v5.5.3软件检查质量,并检查峰图,人工校正后用MEGA X 软件以邻接(Neighbor-joining,NJ)法对所得序列构建系统发育树并计算遗传距离,以Bootstrap 方法对系统发育树进行评估,Bootstrap 次数设置为1 000。依据ISSR 扩增电泳图,选择清晰可辨的电泳条带,分别读取数据,相同迁移率处有带为1,无带为0,建立0/1矩阵,应用NTSYS-pc 2.10软件基于DICE 系数计算遗传相似系数,按照非加权平均法(UPGMA)聚类分析。
2 结果与分析
2.1 18种灵芝菌株的ITS系统发育分析
测序获得18 种灵芝菌株的ITS 序列,经对位排列,人工矫正后长度为576~580 bp,在GenBank中下载 多 孔 菌 目 香 菇 属(Lentinus arcularius,MH142022.1)ITS 序列,长度为648 bp。将其作为外群,将经测序的18种灵芝菌株和GenBank 中已登录的12 条灵芝菌株ITS 序列通过NJ 法构建系统发育树,结果见图1。灵芝属内12 条ITS 序列同一种不同菌株很好地聚在一起;赤芝(Ganoderma lingzhi)与柯蒂斯灵芝(Ganoderma curtisii)聚在一支,支持率为86%;18 种灵芝菌株均与赤芝聚为一类,并可进一步分为2个类群,即类群Ⅰ和类群Ⅱ,支持率为96%。在类群Ⅰ进化分支中可继续分为组a(LZ-2、LZ-13、LZ-15、LZ-16、LZ-18)和组b(LZ-4、LZ-11、LZ-12、LZ-14、LZ-17),支持率为64%;类群Ⅱ被分为组c(LZ-7、LZ-8)和组d(LZ-1、LZ-3、LZ-5、LZ-6、LZ-9、LZ-10),支持率为47%。经各菌株间及组间序列对比发现,18种灵芝菌株各组内各菌株序列相似性为100%,组间序列相似性在99.29%~99.65%。
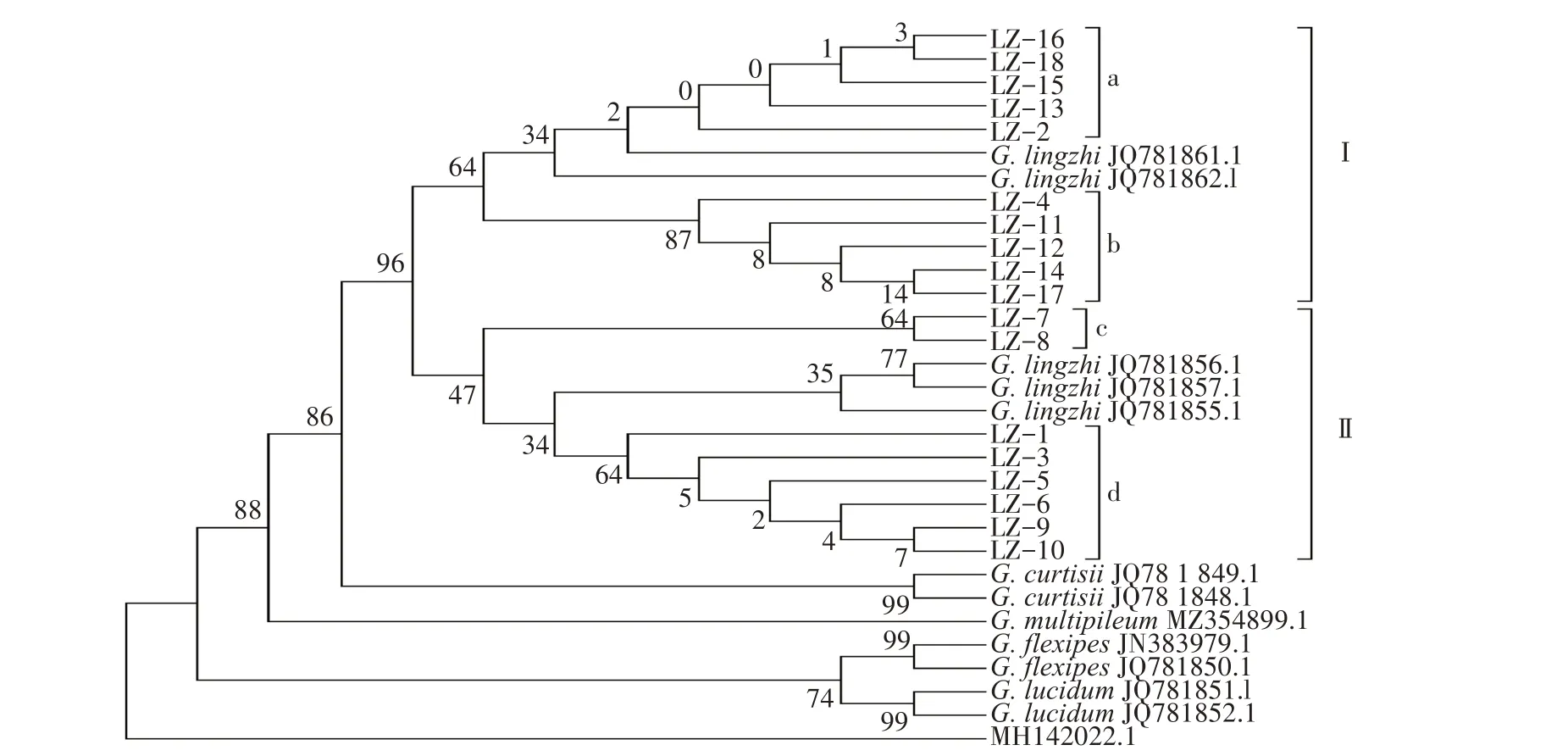
图1 基于18种灵芝菌株ITS序列构建的系统发育树Fig.1 Phylogenetic tree constructed based on the ITS sequences of 18 Ganoderma lucidum strains
2.2 18种灵芝菌株的拮抗试验结果分析
对18 种灵芝菌株共设计拮抗试验153 组,其中有拮抗现象的有122 组,占比79.7%;无拮抗现象的31 组,占比20.3%。多数拮抗现象表现为形成一条沟带。根据ITS 分组结果对组内及组间拮抗现象进行分析(表3)。组a 与组b 之间均有拮抗现象;组a内LZ-13、LZ-15、LZ-18 之间无拮抗现象;LZ-2 与LZ-7 之间无拮抗;LZ-16 与组d 内LZ-3、LZ-9 拮抗不明显,但与组d 内LZ-1、LZ-5、LZ-6、LZ-10 拮抗明显;组b 组内5 个菌株之间均无拮抗现象;组c 与组d 之间均有拮抗现象;组c 内两菌株同样有拮抗现象;组d 内6 个菌株之间均无拮抗现象。拮抗试验结果与ITS 试验结果大体一致。组a 内LZ-13、LZ-15、LZ-18,组b、组d 各组内菌株亲缘关系均较近,可初步判断为同一菌株;LZ-8 与其余各菌株均有拮抗现象,可判断为单一菌株;LZ-2、LZ-7、LZ-16 疑似单一菌株,但需进一步鉴定。通过ITS 扩增和拮抗试验,将18种灵芝菌株分成7组:LZ-13、LZ-15、LZ-18 为一组,组b、组d、LZ-2、LZ-7、LZ-8、LZ-16各自为一组)。
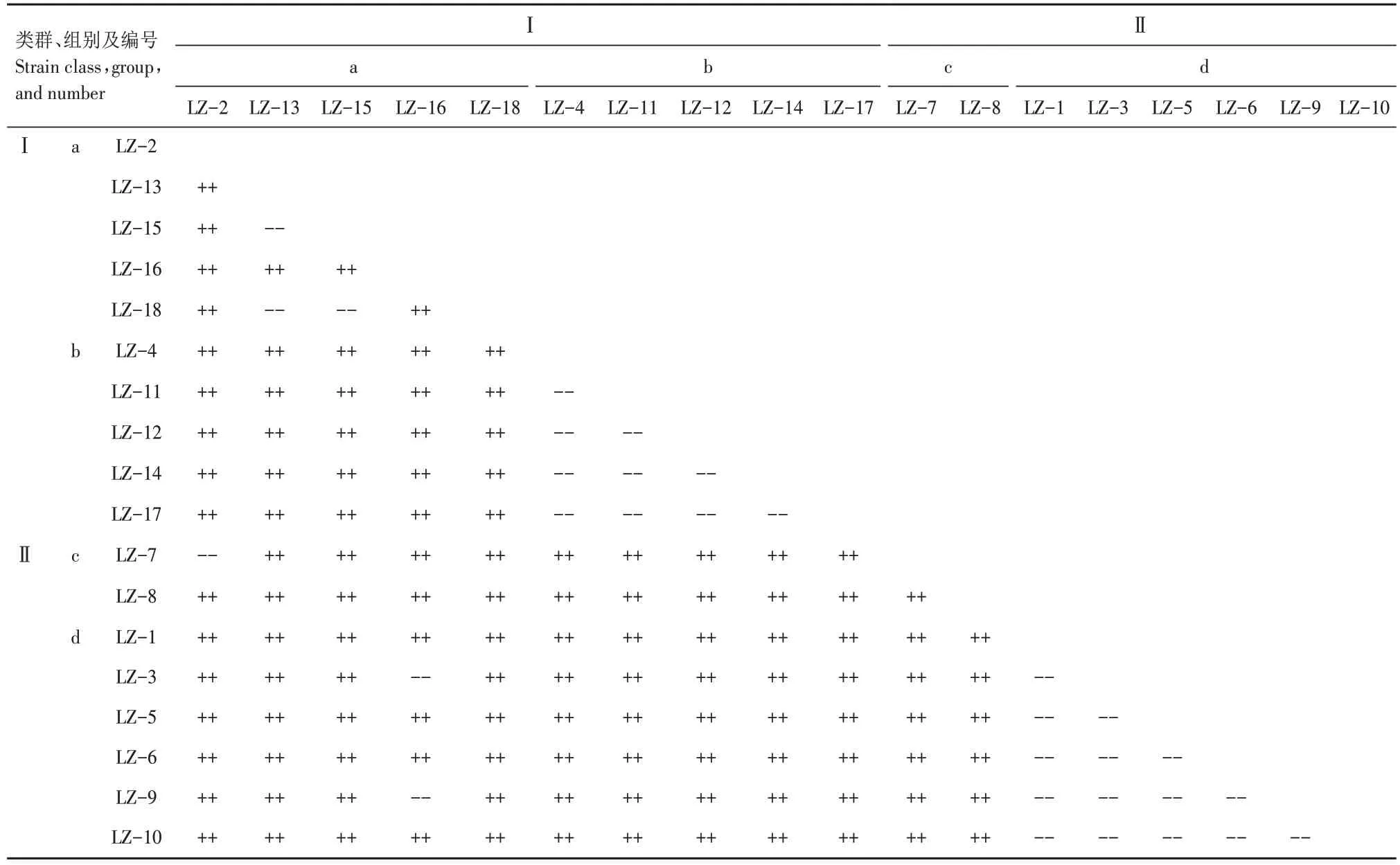
表3 18种灵芝菌株的拮抗试验结果Tab.3 Results of antagonism tests of 18 Ganoderma lucidum strains
2.3 18种灵芝菌株的ISSR遗传多样性分析
2.3.1 ISSR 引物多态性 通过对20 条ISSR 引物进行温度梯度试验后,确定的12 条ISSR 引物的退火温度在43~56 ℃,其在18种灵芝菌株中扩增出91条清晰稳定的条带(表4),DNA 片段大小在100~2 000 bp(图2)。其中,ISSR-6 引物扩增条带最多(12条),ISSR-4和ISSR-12引物扩增条带最少(4条),平均为7.6 条。各ISSR 引物的多态性条带数为3~12条,平均为6.8条,多态性位点百分率为87.2%。
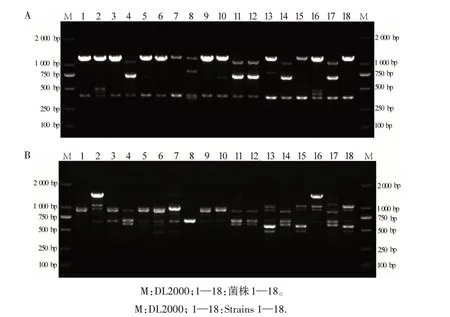
图2 18种灵芝菌株ISSR-3(A)、ISSR-5(B)引物的ISSR扩增图谱Fig.2 ISSR fingerprints generated using primers ISSR-3(A)、ISSR-5(B)of 18 Ganoderma lucidum strains
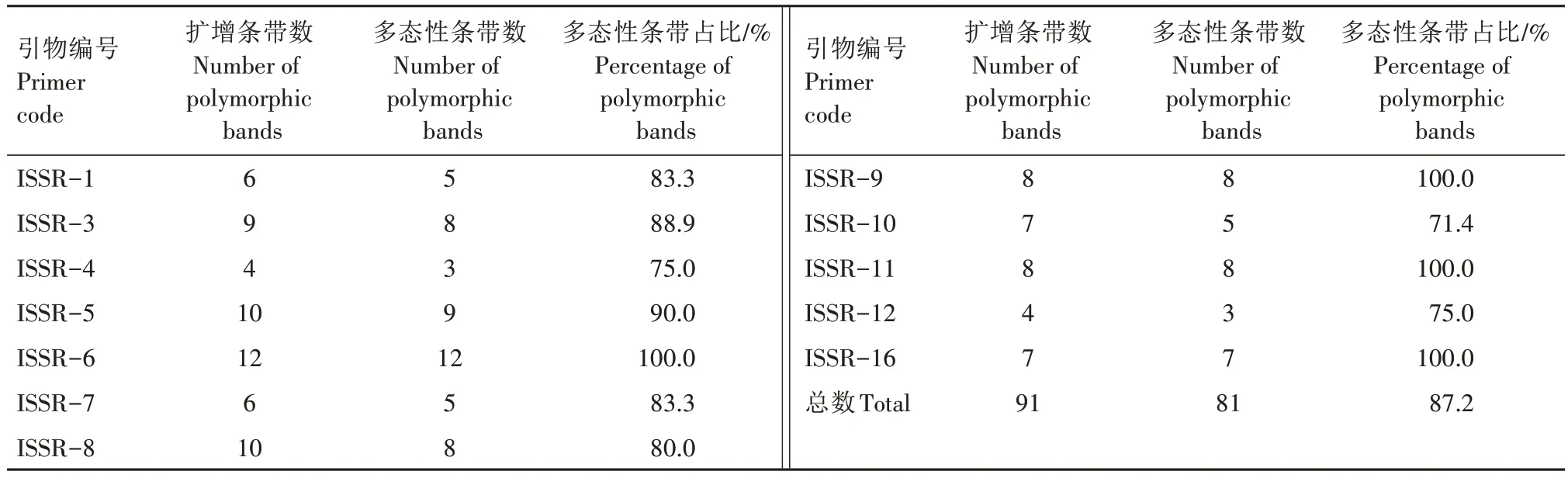
表4 18种灵芝菌株的ISSR引物多态性Tab.4 ISSR primer polymorphisms of 18 Ganoderma lucidum strains
2.3.2 18 种灵芝菌株的聚类分析 18 种灵芝菌株的遗传相似系数在0.382 4~0.974 4,平均为0.652 9,遗传范围差异较大(表5)。其中,LZ-14和LZ-17之间的遗传相似系数最大,为0.974 4,表明两者的亲缘关系最近,遗传差异最小;其次是LZ-11和LZ-12,遗传相似系数为0.970 6;LZ-8和LZ-11的遗传相似系数最小,为0.382 4,表明两者的亲缘关系最远,遗传差异较大。

表5 18种灵芝菌株的遗传相似系数Tab.5 Genetic similarity coefficient of of 18 Ganoderma lucidum strains
基于18 种灵芝菌株的ISSR 遗传多样性结果进行聚类分析,可将18 种菌株分开(图3)。在遗传相似系数约0.550 处,所有菌株被分为2 个类群;在遗传相似系数约0.569 处,所有菌株被分为4 个类群,除LZ-7 外,其余分组与ITS 结果相同。当相似系数约0.679 时,18 种灵芝菌株可分为7 个类群:LZ-2、LZ-7、LZ-8、LZ-16各自成为一个类群,LZ-1、LZ-3、LZ-5、LZ-6、LZ-9、LZ-10 为 一 个 类 群,LZ-13、LZ-15、LZ-18 为一个类群,LZ-4、LZ-11、LZ-12、LZ-14、LZ-17 为一个类群。该聚类结果与基于ITS扩增和拮抗试验分类的结果相同。
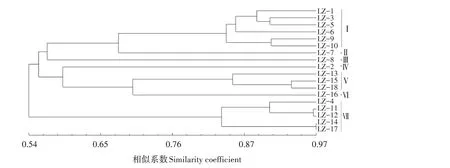
图3 18种灵芝菌株的ISSR聚类分析Fig.3 ISSR cluster diagram of 18 Ganoderma lucidum strains
3 结论与讨论
目前,多种方法可用于食用菌的鉴定及遗传多样性分析[24-25],每种方法所提供的遗传信息不同,因此,对于其遗传关系的反映有所差异,多种方法相结合可以更加准确可靠地反映分析结果。ITS 技术可以有效对灵芝菌株进行分类,研究认为,当序列相似性达99%以上,可认为是同一个种[26]。汤坤鹏[27]利用ITS 技术将18 种野生灵芝菌株分为六大类,其中包括赤芝、紫芝、松杉灵芝等。既往研究中,对于菌株分类常用样本序列进行Blast 对比,但由于18 种灵芝菌株亲缘关系较近,ITS 序列差距较小,且GenBank 数据库中有关灵芝ITS 的序列多、质量参差不齐[8],可靠性有待进一步查证。因此,本研究选择亲缘关系较近的5 个不同种,参考现有文献[28]中分类学已明确的12 条灵芝菌株ITS 序列,与经测序的样本序列构建系统发育树。结果表明,赤芝与柯蒂斯灵芝聚为一类,支持率为86%,其亲缘关系较其他种更近,与前人研究结果相同[28-29];18种灵芝菌株均与赤芝聚为一类,可进一步分为2 个类群,支持率为96%,2 个类群各自独立进化为2 个分支。体细胞的非亲和性往往导致菌丝体间出现明显的分界线,即出现拮抗反应,表现为色素沉淀、形成一条拮抗线、沟带或隆起[30]。拮抗反应操作简单、便捷,结果直观,是14 种食用菌新品种DUS 测试指南中的测试项目之一。研究认为,2 个菌株对峙培养,若没有拮抗反应,不能判定为同一菌株,需进一步鉴定[20],拮抗试验常用于辅助其他手段进行菌株的鉴定及分析亲缘关系[13,31]。崔筱等[32]在对河南主产区香菇进行种质资源鉴定及遗传多样性分析时认为,ITS+拮抗试验法可以明确区分遗传距离的差异及亲缘关系的远近,与本研究结果相似,通过ITS+拮抗法将18种灵芝菌株分为7个类群。
分子标记是以DNA 多态性为基础反映遗传多样性的一种手段。王艳等[33]采用ISSR 和相关序列扩 增 多 态 性(Sequence related amplified polymorphism,SRAP)综合分析灰树花遗传多样性时发现,ISSR扩增检测到的位点数比SRAP多,多态性比率比SRAP 高,相比于其余分子标记手段,其具有多态性丰富、操作简单、无需提前知道基因组相关序列信息等优点[29]。通过ISSR 分子标记技术,陈小敏等[34]将四川省12 个野生灵芝菌株在相似系数为0.54 时聚为两大类,张彬彬等[35]将河南省30 个野生灵芝菌株在相似系数为0.728 时分为六大类。王锦锋等[36]利用拮抗、ITS 和随机扩增多态性(Random amplified polymorphic DNA,RAPD)技术对21 株灵芝菌株进行研究,认为3种方法结果一致,且能准确鉴定其亲缘关系。本研究采用ITS+拮抗试验法和ISSR 技术相结合对吉林地区18 种栽培灵芝菌株进行鉴定及遗传多样性分析。基于ISSR结果,在遗传相似系数约0.550 时,分为2 个类群;在遗传相似系数约0.569 时,分为4个类群。ISSR 分类结果表明,其在遗传相似系数约0.679 处与ITS+拮抗试验法分类结果一致。本研究中,18种灵芝菌株具有丰富的遗传多样性。

