2型猪链球菌N-乙酰氨基葡糖-6-磷酸脱乙酰酶基因的克隆表达及酶活性测定
钱思彤,龚秀芳,唐慧娴,王依,潘秀珍,王长军
1.中国药科大学 药学院,江苏 南京 211198;2.南京军区 军事医学研究所,江苏 南京 210002
研究报告
2型猪链球菌N-乙酰氨基葡糖-6-磷酸脱乙酰酶基因的克隆表达及酶活性测定
钱思彤1,2,龚秀芳1,2,唐慧娴1,2,王依2,潘秀珍2,王长军1,2
1.中国药科大学 药学院,江苏 南京 211198;2.南京军区 军事医学研究所,江苏 南京 210002
目的:克隆表达2型猪链球菌N-乙酰氨基葡糖-6-磷酸脱乙酰酶(NagA)的编码基因,并测定其酶活性。方法:根据GenBank中05ZYH33基因组序列设计引物,PCR扩增NagA(SSU05_1259)基因,将其克隆到pET28a载体中,构建重组质粒pET28a:NagA,转化至大肠杆菌BL21,筛选阳性转化子进行IPTG诱导表达,产物通过SDS-PAGE与质谱鉴定;利用Ni亲和层析柱对表达产物进行纯化,获得NagA重组蛋白后测定其酶活性。结果:在大肠杆菌中高效表达了NagA基因,重组表达的NagA相对分子质量约为43×103,其酶促反应最适温度为37℃,最佳反应时间为30 min,最适反应pH值为7.5,最佳底物浓度为13 mmol/L。2型猪链球菌NagA的体外酶活为124 U/mL,酶比活为78 U/ mg。结论:在原核系统中表达了NagA基因,获得的NagA蛋白具有良好的酶学活性。
2型猪链球菌;N-乙酰氨基葡糖-6-磷酸脱乙酰酶;原核表达;酶活性
猪链球菌(Streptococcus suis,SS)是一种重要的人兽共患病病原,属于链球菌属,是革兰阳性细菌,可引起猪脑膜炎、关节炎、肺炎、败血症甚至急性死亡[1-2]。目前,世界范围内发生的该菌感染人的病例已逾400例,70余患者死亡[1]。2型猪链球菌(SS2)是猪链球菌35种血清型中致病力最强、临床检出率最高、呈全球性分布的血清型,是重要的人兽共患病病原[3]。我国江苏海安和四川资阳分别于1998、2005年暴发SS2感染人的公共卫生事件,患者中出现了链球菌中毒性休克综合征(streptococcal toxic shock syndrome,STSS),引起全世界的广泛关注[3-4]。
猪链球菌的不同血清型菌株的致病性差异较大,这与其复杂的致病机制密切相关,主要涉及毒力因子的黏附、入侵、扩散等方面,目前尚未完全阐明,报道较多的相关毒力因子有荚膜多糖(capsular polysaccharide,CPS)、胞外因子(exracel⁃lular factor,EF)、溶菌酶释放蛋白(muraminidase released protein,MRP)、溶血素(suilysin,SLY)等,可能在感染致病的不同阶段先后或协调发挥作用[5-7]。蛋白酶是重要的功能分子,在细菌修饰、活化与代谢中发挥重要功能,也可参与细菌侵袭致病过程[8]。已发现N-乙酰氨基葡萄糖-6-磷酸的脱乙酰作用是脂多糖合成和细胞壁再生的重要环节,葡萄糖胺-6-磷酸是GalNAc/GalN代谢通路的正常中间体,同时葡萄糖胺-6-磷酸还可通过NagS转化为果糖-6-磷酸。在革兰阴性菌如大肠杆菌中,该反应也是脂多糖合成和细胞壁氨基糖回收的重要途径,负责催化该反应的酶就是N-乙酰氨基葡萄糖-6-磷酸脱乙酰酶(N-acetylglu⁃cosamine-6-phosphate deacetylase,NagA)[9-11]。现已发现NagA在枯草芽孢杆菌和大肠杆菌的致病性中发挥重要作用[12-14],但SS2是否表达该酶以及其是否参与了细菌的致病过程尚未见报道。本课题组在对SS2中国强毒株05ZYH33进行全基因组测序和功能注释时,在基因组中发现了一个与枯草芽孢杆菌和大肠杆菌NagA编码基因同源性较高的类似物,进而构建了适宜的原核表达系统对该基因进行了克隆表达,纯化其表达产物,并进一步分析其酶活性,为研究NagA在SS2糖代谢以及猪链球菌致病过程中可能发挥的作用奠定了基础。
1 材料与方法
1.1 材料
SS2菌株05ZYH33、表达质粒pET-28a由本实验室保存;受体菌大肠杆菌DH5α及BL21购自全式金生物技术有限公司;pMD-19T质粒、PCR扩增试剂盒、限制性内切酶XhoⅠ和BamHⅠ、DNA marker购自TaKaRa有限公司;DNA割胶回收试剂盒购自Promega公司;蛋白质marker购自晶美公司;NagA反应底物N-乙酰氨基葡萄糖-6-磷酸钠盐(GlcNAc-6-P)购自Sigma-Alorich生物工程有限公司;紫外分光光度计UV2100为尤尼柯公司产品。
1.2 引物设计
根据05ZYH33基因组中NagA基因序列设计上游引物(5'-GAGGGATCCATGGTTATAACCAGT TT-3',下划线部分为BamHⅠ酶切位点)、下游引物(5'-ATACTCGAGACCAAATCGTTTCACG-3',下划线部分为XhoⅠ酶切位点),由上海赛百盛公司合成。
1.3 NagA基因的扩增
常规PCR扩增目的基因,25 μL反应体系包括05ZYH33基因组DNA 1 μL,上、下游引物各0.5 μL,dNTP混合液2 μL,10×PCR缓冲液(加Mg2+)2.5 μL,Taq DNA聚合酶0.2 μL,dd H2O 18.3 μL。
反应参数:95℃ 3 min预变性;94℃ 30 s、54℃ 1 min、72℃ 1 min,30个循环;72℃延伸10 min。1%琼脂糖凝胶电泳分析PCR产物。
1.4 pMD19-T克隆质粒的构建及鉴定
用DNA胶回收试剂盒回收琼脂糖凝胶中的目的基因片段,连接至pMD19-T载体,连接产物转化至大肠杆菌Trans-T1感受态细胞,挑选阳性克隆,提质粒;质粒pMD19-T:NagA经PCR和XhoⅠ、BamHⅠ双酶切鉴定正确后,送华大公司测序,分析结果。
1.5 pET28a:NagA质粒的构建
将1.4构建的pMD19-T:NagA质粒和pET28a载体分别用XhoⅠ和BamHⅠ内切酶酶切,经1%琼脂糖凝胶电泳分离,割胶回收NagA目的片段和线性化的pET28a片段;用DNA高效连接酶于16℃连接2 h,连接产物转化大肠杆菌DH5α,挑选阳性单克隆,提质粒;pET28a:NagA质粒经PCR、双酶切鉴定正确后,送华大公司测序鉴定,分析结果。
1.6 重组质粒pET28a:NagA的诱导表达
1.6.1 重组蛋白NagA表达形式鉴定 将重组质粒pET28a:NagA转化大肠杆菌BL21,经IPTG于37℃诱导表达4 h,收集菌体于冰浴中超声波破碎,离心后取上清和沉淀分别进行SDS-PAGE,观察分析是否有目的蛋白的表达。
1.6.2 重组蛋白NagA的纯化 离心收集表达菌液破碎后的上清,用0.22 μm滤膜过滤,过滤后的上清液与用PBS平衡过的Ni离子亲和层析柱填料螯合,冰浴2 h,用90 mmol/L咪唑洗脱并收集目的蛋白,10%SDS-PAGE鉴定目的蛋白的相对分子质量。
1.7 目标蛋白的质谱分析
从SDS-PAGE胶上切取含有目标蛋白质的条带,经胶内酶解后用Q-TOF质谱仪分析,提取质谱数据,用Data Analysis软件标峰后进行MAS⁃COT搜索,得到重组蛋白的LC-MS/MS报告单,从而判定是否为NagA。
1.8 NagA酶学性质测定
1.8.1 酶活性测定 37℃时每分钟消耗1 μmol GlcNAc-6-P生成1 μmol产物的NagA量为一个酶单位(U)。GlcNAc-6-P于215 nm处酰胺基团有紫外吸收,水解反应后吸收消失,故可以通过1 min内底物D215nm值的减少量来确定参与反应的底物量,确定其酶活性。最后确定的检测方法为:在总体积为 1mL的含 13mmol/L底物GlcNAc-6-P的Tris-HCl缓冲液(50 mmol/L Tris-HCl,pH7.5)体系中,加入纯化的重组NagA 1.6 μg,37℃反应30 min,于沸水中5 min终止反应,分别测量反应前后的D215nm值,通过D215nm值的减少量计算参加反应的底物量。按下式计算酶活性:
酶活性(U/mL)=(ΔD215nm×1000)/(0.331×30)其中,ΔD215nm为反应前后的吸光度差值,1000为将1 μL稀释酶液的酶活性折算为1 mL的酶活性,30为反应时间(30 min),0.331为标准曲线系数。
1.8.2 GlcNAc-6-P吸光度标准曲线 据文献[15],GlcNAc-6-P的吸收峰为200~300 nm,在215 nm处有特征吸收。配制浓度分别为0、0.5、1、1.5、2、2.5 mmol/L的GlcNAc-6-P溶液,定容至1 mL,各取200 μL测定D215nm值(以下所有吸光度值均为测量3次后的平均值),制作GlcNAc-6-P吸光度标准曲线。
1.8.3 最适温度 为确定温度对NagA活性的影响,在pH7.5、反应时间30 min时分别测定25℃、30℃、37℃、42℃、50℃、60℃和70℃时的酶活性。
1.8.4 最适反应时间 为确定酶促反应的最适反应时间,在37℃、pH7.5条件下,分别测定反应10、15、20、25、30、35、40、45、50、60及90 min时的酶活性。
1.8.5 最适pH值 为确定pH值对NagA活性的影响,在37℃、反应时间30 min时,测定pH值分别为3、5、6、6.5、7、7.5、8、8.5、9、10时的酶活性。
1.8.6 底物浓度对酶活的影响 为探索底物浓度对NagA活性的影响,在pH7.5、37℃条件下,分别将底物浓度设为1、3、5、7、9、11、13、15、20 mmol/ L,观察该酶的活性。
2 结果
2.1 2型猪链球菌NagA基因序列分析
以05ZYH33菌株为模板,PCR扩增得到约1200 bp的目的条带,测序结果显示序列全长1191 bp,编码397个氨基酸残基,读框正确无误。进行BLAST比对发现NagA蛋白在猪链球菌不同菌株中高度保守,氨基酸序列相似性在90%以上,与牛链球菌(S.ovis)、嗜热链球菌(S.ther⁃mophilus)、枯草芽孢杆菌(Bacillus cereus)、S.merio⁃nis等的氨基酸序列相似性分别为85%、63%、61%和61%,其氨基酸序列比对结果如图1,图2为扩增片段的电泳图谱。
2.2 重组表达质粒pET28a:NagA的构建和鉴定
pET28a:NagA质粒经XhoⅠ和BamHⅠ双酶切后进行1%琼脂糖凝胶电泳,结果如图3,pET28a: NagA经双酶切后得到明显的2条带,其中1200 bp左右的小片段为目的基因NagA,3500 bp左右的大片段为pET28a线性化载体,而pET28a空载体经双酶切得到大于4500 bp的大片段。测序结果显示,目的基因与GenBank中SS2中国强毒株05ZYH33的NagA基因的序列一致,证明重组质粒构建成功。

图1 NagA蛋白的氨基酸序列比对图
2.3 NagA的表达及纯化
2.3.1 重组质粒pET28a:NagA的诱导表达鉴定诱导表达的重组菌经超声波裂解后离心,进行SDS-PAGE分析(图4),发现目的蛋白大部分存在于沉淀中,约占60%,上清中含有少部分,约占30%。显示目的蛋白主要以包涵体形式存在。
2.3.2 重组蛋白NagA的纯化 选取上清中可溶性蛋白,经10%SDS-PAGE,在相对分子质量约43×103处出现新生蛋白条带,大小与预测结果一致。表达产物经His亲和层析柱纯化,电泳结果显示纯化产物为单一特异性条带(图5)。
2.4 NagA重组蛋白的质谱分析

图2 NagA基因的PCR扩增

图3 pET28a:NagA重组质粒的XhoⅠ、BamHⅠ双酶切鉴定

图4 NagA重组蛋白的可溶性表达鉴定
质谱结果显示NagA重组蛋白与预测的NagA序列相似度较高,结果如下,其中划线部分为氨基酸序列完全一致的区域:

2.5 重组NagA的酶活性测定
2.5.1 GlcNAc-6-P吸光度标准曲线 215 nm下绘制的底物标准曲线如图6。
2.5.2 温度对酶活性的影响 在一定条件下,一种酶只在特定的反应温度下表现出最大的活性,该温度即为最适温度。在pH7.5、反应时间30 min的条件下,当温度低于37℃时,酶活性随温度的升高而升高,37℃时酶活性达到峰值,此后酶活性保持在一个较低水平,当反应温度高于60℃时酶活性急剧下降(图7)。因此,该酶最适反应温度约为37℃。

图5 SDS-PAGE检测蛋白表达和纯化

图6 GlcNAc-6-P吸光度标准曲线
2.5.3 反应时间对酶活性的影响 在pH7.5、37℃条件下分别检测不同反应时间点的酶活性,结果如图8。在10~30 min内,随着反应时间的延长,NagA酶促反应速度显著增加,当反应时间为30 min时反应速度达到最大值,反应时间超过30 min时反应速度降低,反应呈低水平抑制状态,推测NagA酶促反应产物之一的N-氨基葡糖-6-磷酸(GlcN-6-P)可能是NagA的竞争性抑制剂[15],NagA酶的脱乙酰作用可能受其产物浓度的影响,故SS2中NagA酶的最适反应时间约为30 min。见图8。
2.5.4 pH值对酶活性的影响 同一种酶在不同pH值环境中所表现的活性不同,在特定条件下,酶显示最大活性的pH值即为该酶的最适pH值。在37℃、反应时间30 min的条件下,测定不同pH值对NagA酶活性的影响,发现该酶在酸性条件下的活性高于碱性条件,且在pH7.5时显示最高活性(图9)。
2.5.5 底物浓度对酶活的影响 测定pH7.5、37℃时酶活性与底物浓度的关系,结果如图10。当底物浓度为1~13 mmol/L时,NagA酶活性随底物浓度的增加而增加,底物浓度为13 mmol/L时酶促反应达到饱和。考虑到相关成本因素,默认当底物浓度为13 mmol/L时酶活性最大。
2.5.6 酶活性测定 根据底物GlcNAc-6-P吸光度标准曲线及NagA活计算公式,可得纯化后的2型猪链球菌NagA的体外酶活为124 U/mL。通过One-Drop测定纯化后的重组NagA蛋白含量为1.6 mg/mL,计算可得酶比活为78 U/mg。

图7 反应温度与酶活性的关系
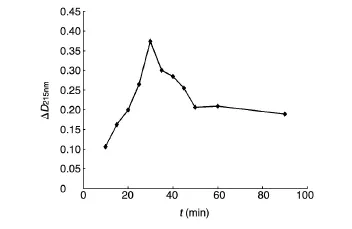
图8 反应时间与酶活性的关系
3 讨论
细菌可以有效利用氨基糖合成细胞壁中的肽聚糖和细胞膜中的脂多糖,同时氨基糖也是细菌生长与增殖中良好的碳源与氮源[15]。当细菌生长环境中存在氨基糖时,会自发诱导产生与氨基糖类吸收和代谢相关的酶。N-乙酰氨基葡糖-6-磷酸脱乙酰酶是氨基糖代谢过程中一种重要的酶,可以将N-乙酰氨基葡糖-6-磷酸水解为N-氨基葡糖-6-磷酸和醋酸[16],该步骤是N-乙酰氨基葡糖同化作用和氨基糖核苷酸生物合成中的第一个关键步骤[17]。本课题组在进行05ZYH33全基因组注释时发现了可能编码NagA的基因SSU1259,其推导的氨基酸序列与牛乳链球菌及枯草芽孢杆菌中的NagA氨基酸序列具有较高的同源性,推测其编码NagA蛋白。基于此,本研究克隆表达了编码该酶的目的基因,分析了重组蛋白的酶学活性与功能,为确定其在SS2可能的致病性中的作用以及相关疫苗的研究奠定了基础。

图9 pH值与酶活性的关系

图10 底物浓度与酶活性的关系
本研究克隆表达的中国强毒株05ZYH33中NagA基因全长1191 bp,编码由397个氨基酸残基组成的蛋白,质谱检测蛋白的相对分子质量约43×103。经原核表达及His亲和层析获得高纯度的NagA重组蛋白,进一步酶活性检测表明SS2的NagA与其他已知来源的NagA蛋白既具有相似的酶学功能,也具有其独有的酶学特性。SS2来源的NagA酶的最适反应温度为37℃,而大肠杆菌[15]和栖热菌Thermus caldophilus[16]的NagA酶的最适反应温度分别为30℃、70℃[16],猜测可能与SS2是人猪共患病病原有关,其最适反应温度与宿主温度接近。SS2的NagA最适pH值范围较窄,为7~7.5,偏离上述条件时酶活性迅速下降;而大肠杆菌NagA酶的最适反应pH值为7.5,T.caldophilus中NagA的最适反应pH值为7.0[15-16]。上述不同来源的NagA酶在温度和pH值敏感性上的差异可能是微生物适应各自宿主环境的表现,也可能是自然选择的结果[18]。此外,不同来源的NagA酶的最适反应时间与最适底物浓度也不尽相同,本研究中NagA的最适反应时间是30 min,随着反应时间的增加,酶促反应呈低水平抑制状态,推测NagA酶促反应产物之一的GlcN-6-P可能是NagA的竞争性抑制剂。当底物浓度达到一定值后,酶促反应达到饱和,其反应速率不再随底物浓度的增加而增加。
综上所述,我们克隆表达了SS2中的NagA基因,获得了纯度较高的N-乙酰氨基葡糖-6-磷酸脱乙酰酶蛋白,对其酶活特性进行了系统分析。研究结果为进一步分析NagA在2型猪链球菌糖代谢及感染致病过程中的作用奠定了基础。
[1]Lun Z R,Wang Q P,Chen X G,et al.Streptococcus suis:an emerging zoonotic pathogen[J].Lancet Infect Dis,2007,7(3):201-209.
[2]Gottschalk M,Segura M,Xu J.Streptococcus suis in⁃fections in humans:the chinese experience and the sit⁃uation in north America[J].Anim Health ResRev, 2007,8(1):29-45.
[3]Tang J Q,Wang C J,Feng Y J,et al.Streptococcal toxic shock syndrome caused by Streptococcus suis se⁃rotype 2[J].PLoS Med,2006,3:e151.
[4]Sriskandan S,Slater J D.Invasive disease and toxic shock due to zoonotic Streptococcus suis:an emerging infection in the east[J].PLoS Med,2006,3(5):e187.
[5]Thiebaut R,Leproust S,Chen G,et al.Effectiveness of prenatal treatment forcongenital toxoplasmosis:a metaanalysis ofindividualpatients,data[J].Lancet, 2007,369(9556):115-122.
[6]Innes E A,Vermeulen A N.Vaccination as a control strategy Naginst the coccidial parasites Eimeria,Toxo⁃plasma and Neospora[J].Parasitology,2006,133:S145-S168.
[7]Jiang W W,Baker H J,Smith B F.Mucosal immuni⁃zation with Helicobacter,CpG DNA,choleratoxin is protective[J].Infect Immun,2003,71(1):40-46.
[8]Maeda H.Role of microbial proteases in pathogenesis [J].Microbiol Immunol,1996,40(10):685-699.
[9]Barnhart M M,Lynem J,Chapman M R.GlcNAc-6-P levels modulate the expression of Curli fibers by Escherichia coli[J].J Bacteriol,2006,188:5212-5219.
[10]Park J T.Identification of a dedicated recycling path⁃way for anhydro-N-acetylmuramic acid and N-acetyl⁃glucosamine derived from Escherichia colicellwall murein[J].J Bacteriol,2001,183:3842-3847.
[11]Jaeger T,Mayer C.N-acetylmuramic acid-6-phos⁃phate lyases:role in cell wall metabolism,distribu⁃tion,structure,and mechanism[J].Cell Mol Life Sci, 2007,65:928-939.
[12]Plumbridge J A.Induction of the nag regulon of Esch⁃erichia coli by N-acetyl-D-glucosamine and glucos⁃amine:role of the cyclic AMP-catabolite activator pro⁃tein complex in expression of the regulon[J].J Bacteri⁃ol,1990,172:2728-2735.
[13]Freese E B,Cole R M,Klofat W,et al.Growth,spor⁃ulation,and enzyme defects of glucosamine mutants of Bacillus subtilis[J].J Bacteriol,1970,101:1046-1062.
[14]Mobley H L T,Doyle R J,Streips U N,et al.Trans⁃port and incorporation of N-acetyl-D-glucosamine in Bacillus subtilis[J].J Bacteriol,1982,150:8-15.
[15]Souza,J M,Plumbridge J A,Calcagno M L.N-acetyl⁃glucosamine-6-phosphate deacetylase from Escherichia coli:purification and molecular and kinetic character⁃ization[J].Arch Biochem Biophys,1997,340:338-346.
[16]Shin H J,Kim M,Lee D S.Purification and charac⁃terization of N-acetylglucosamine-6-phosphate deacety⁃lase from Thermus caldophilus[J].J Biosci Bioeng, 1999,88(3):319-322.
[17]Yadav V,Panilaitis B,Shi H,et al.N-acetylglucos⁃amine-6-phosphate deacetylase(nagA)is required for N-acetyl glucosamine assimilation in gluconacetobacter xylinus[J].PLoS One,2011,6(6):e18099.
[18]张凤玉,胡丹,龚秀芳,等.2型猪链球菌β-半乳糖苷酶基因的克隆表达及酶活性测定[J].中国生物工程杂志,2014,34(2):39-44.
Gene Cloning,Expression and Identification of the NAcetylglucosam ine-6-Phosphate Deacetylase of Streptococcus suis serotype 2
QIAN Si-Tong1,2,GONG Xiu-Fang1,2,TANG Hui-Xian1,2, WANG Yi2,PAN XIU-Zhen2,WANG Chang-Jun1,2*
1.School of Pharmacy,China Pharmaceutical University,Nanjing 211198;
2.Institute of Military Medical Sciences,Nanjing Military Region,Nanjing 210002;China
Objective:To clone and prokaryotically express the gene encoding N-acetylglucosamine-6-phosphate deacetylase(NagA)of Streptococcus suis serotype 2 and to measure the enzymatic activity of the recombinant pro⁃tein.Methods:The gene encoding NagA was amplified by PCR from the template S.suis 05ZYH33 genomic se⁃quence,and was cloned into vector pET28a to obtain the recombinant plasmid pET-28a:NagA,then it was trans⁃formed into E.coli BL21.The recombinant NagA protein was induced to express by IPTG,and analyzed with SDSPAGE and LC-MS/MS,purified by Ni affifnity chromatography,followed by enzymatic activity measurement.Re⁃sults:NagA gene(SSU05_1259)was expressed in E.coli with Mrabout 43×103.The optimum temperature,action time,pH and the substrate concentration of the purified NagA protein were 37℃,30 min,7.5 and 13 mmol/L,re⁃spectively.Enzymatic activity of NagA was 124 U/mL,specific activity was 78 U/mL.Conclusion:The NagAgene has been expressed in prokaryotic system,and the purified recombinant protein has the best enzymatic activi⁃ty in optimum experimental condition.
Streptococcus suis seroptype 2;N-acetylglucosamine-6-phosphate deacetylase;prokaryotic expres⁃sion;enzymatic activity
Q78
A
1009-0002(2017)02-0069-08
10.3969/j.issn.1009-0002.2017.02.001
2016-11-18
国家自然科学基金(81471920,81501725);江苏省自然科学青年基金(BK20140096);南京军区军事医学科技创新课题(ZX39)
钱思彤(1992-),女,硕士研究生
王长军,(E-mail)science2008@hotmail.com
*Corresponding anthor,E-mail:science2008@hotmail.com

